花生4CL基因家族鉴定及对干旱与盐胁迫响应分析--文献精读157
花生4CL基因家族鉴定及对干旱与盐胁迫响应分析

摘要
目的 分析花生4-香豆酸:CoA连接酶(4-coumarate:CoA ligase, 4CL)基因家族成员基本特性及其对干旱和盐胁迫的响应,为培育花生耐旱抗盐品种提供重要的目标基因。 方法 通过HMM文件及NCBI CDD和Pfam数据库在全基因组水平鉴定花生4CL基因家族成员;利用ExPASy-ProtParam工具进行蛋白理化性质分析;通过MEGA7及itol工具进行系统进化分析;通过MEME及NCBI中的CD-search工具进行蛋白保守基序和保守结构域分析;通过PlantCARE及TBtools进行启动子元件分析及可视化;通过RNA-seq数据及RT-qPCR分析花生4CLs转录水平变化。 结果 借助花生Tifrunner基因组参考数据,鉴定到56个花生4CL基因,氨基酸长度介于239‒1 208之间,pI介于5.5‒9.22之间,蛋白脂肪系数介于80.2‒103.13之间,不稳定性指数在25.51‒48.79之间,GRAVY值在-0.367‒0.139之间;花生A与B基因组的20条染色体上,Ah4CLs基因不均匀地分布,5和15号染色体Ah4CLs基因密度最高;同一个聚类分支,Ah4CLs具有相似的保守基序组成及相似的内含子‒外显子分布结构,外显子数量在1‒18之间;Ah4CLs启动子区域富含光、非生物胁迫、激素及生长发育响应元件;Ah4CLs表达具有组织特异性,在根、花及种子中表达量较高;在ABA、盐与干旱胁迫处理下,部分Ah4CLs转录水平显著增加,特别是Ah4CL28在ABA、干旱及盐处理下均明显转录上调,这些基因在花生应对非生物胁迫中可能发挥着重要作用。 结论 鉴定到的56个花生4CL基因家族成员有不同结构与特性,在部分基序及结构域上具有保守型,Ah4CLs在影响花生生长发育的同时,还参与非生物胁迫响应。
花生(Arachis hypogaea L.)作为重要的油料及经济作物,是国家食用油供给的重要支柱[1]。干旱、半干旱以及盐渍化的土壤环境是花生种植过程中面临的主要非生物胁迫,使得花生品质与产量受到较为严重的影响[2-5]。抗逆优质花生品种的选育是缓解胁迫的重要方式之一。苯丙烷类代谢途径是植物正常生长所必需的,大量次生代谢产物的前体都由这一途径产生,在植物生长发育以及外部环境胁迫中发挥重要功能[6]。
4CL是苯丙烷类代谢通路中的重要枢纽蛋白,催化肉桂酸及其甲氧基或者羟基衍生物反应生成对应的CoA酯[7]。这些辅酶A酯是植物酚类次级产物的重要中间体,如木质素单体和类黄酮等[8]。已有的研究表明,4CL蛋白序列中存在一些保守的多肽基序,特别是BOX I(SSGTTGLPKGV)和BOX II(GEICIRG)[7],BOX I代表腺苷单磷酸(AMP)结合域,这段基序在所有腺苷酸形成酶家族成员中高度保守[9]。BOX II并不直接参与催化反应,但是这一保守序列的缺失会导致4CL酶活性的几乎完全丧失[10]。随着许多物种基因组测序的完成,许多编码腺苷酸形成酶的基因被发现与已鉴定的4CL基因密切相关[11],但它们大多数的生化功能仍不清楚。在拟南芥(Arabidopsis thaliana (L.) Heynh.)中,发现有9个基因与4CLs编码基因相似,被不同的研究人员鉴定归类为4CL或类4CL基因[12-13]。
已有报道表明,许多4CLs在非生物胁迫中发挥着重要的功能,经历胁迫时,其表达量会发生显著变化[14-15]。白光、紫外线照射以及外源ABA和PEG处理,均可刺激马铃薯(Solanum tuberosum L.)St4CL6和St4CL8的表达上调,同时抑制St4CL5的表达[16]。Gh4CL7沉默棉花(Gossypium hirsutum L.)株系对干旱胁迫表现出较强的敏感性,而Gh4CL7过表达拟南芥植株对干旱的耐受性增强[17]。盐胁迫后,杜仲(Eucommia ulmoides Oliv.)中多数Euc4CLs的表达量显著增加,尤其是Euc4CL9、Euc4CL17和Euc4CL27[18]。由此可见,在非生物胁迫响应与调控方面,4CL基因在不同物种中相对保守。花生4CL基因家族尚未进行比较系统的全基因组鉴定与分类,在非生物胁迫尤其是干旱与盐胁迫中的功能尚不明确。
本研究借助花生品种Tifrunner基因组参考数据进行花生4CL基因家族的鉴定,并对其进化关系、蛋白特性以及表达模式等方面进行分析,同时借助转录组数据以及RT-qPCR分析鉴定受干旱与盐胁迫响应的Ah4CLs,进而为抗旱耐盐花生新品种的培育提供重要的基因位点,借助基因工程手段对这些位点进行改善或许可以为花生抗逆性的提高以及花生产业化提供重要的帮助。
1 材料与方法
1.1 材料
采用试验材料为花生品种狮头企[19],该品种已经进行了全基因组组装与测序,并公布了参考基因组数据,便于进行引物合成与基因表达分析。用0.5%的次氯酸钠溶液将花生种子进行消毒灭菌,并用无菌水清洗,随后在无菌双层滤纸上进行萌发,光照/黑暗时间为16 h/8 h,温度为20 ℃,相对湿度为60%,将萌发且生长一致的幼苗进行种植,待长到3‒4叶期时,将花生幼苗的根浸入Hoagland培养液中进行处理,参照已报道的文献,用20% PEG6000模拟干旱胁迫[20],200 mmol/L NaCl模拟盐胁迫[21],分别在处理0、6、12、24和48 h时取材花生展开叶、幼叶以及根部,用液氮速冻后,放到-80 ℃进行保存备用。
1.2 方法
1.2.1 花生4CL基因家族成员鉴定与蛋白理化性质分析
花生Tifrunner品种基因组已被用于多项分子研究,具有更好的连续性与完整性,其全基因组文件、基因组注释文件以及蛋白序列文件在NCBI(https://www.ncbi.nlm.nih.gov/datasets/taxonomy/3818/)中下载获得。通过HMM文件PF00501和PF13193的筛选获得了花生中4CL候选成员,并通过NCBI CDD和Pfam数据库进一步分析候选成员的蛋白结构域。最终将在CDD数据库中包含4CL结构域和AFD_class_I超家族结构域以及在Pfam数据库中包含AMP-binding和AMP-binding_c结构域的蛋白鉴定为花生4CL基因家族成员。利用ExPASy上的ProtParam(https://web.expasy.org/protparam/)在线工具预测花生4CL蛋白的理化性质。
1.2.2 花生4CL基因家族成员染色体分布及进化分析
花生4CL基因家族的染色体分布位置基于Tifrunner基因组注释文件,候选Ah4CL基因在花生染色体上的物理分布借助TBtools[22]工具进行注释。为了进一步研究的方便,花生4CL基因按照染色体位置进行重新命名。利用MEGA7(https://www.megasoftware.net/)采用邻接法(neighbor-joining, NJ)将鉴定的56个Ah4CL蛋白与拟南芥4CL蛋白进行多序列比对和系统发育树的构建,Bootstrap值设为1 000。最后利用itol(https://itol.embl.de/)在线工具对系统发育树进行美化。
1.2.3 花生4CL基因家族蛋白保守基序、结构域及基因结构分析
花生Tifrunner基因组注释文件辅助进行基因结构分析。为了分析花生4CL蛋白保守域,使用NCBI中的CD-search工具(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)进行搜索,将得到的hitdata.txt文件进行进一步结构域分析。利用MEME版本5.5.5(https://meme-suite.org/meme/tools/meme)进行4CL家族蛋白的保守基序分析,将获得的meme.xml文件用于进一步基序分析。最后使用TBtools工具将上述分析结果进行可视化。
1.2.4 花生4CL基因家族成员共线性及启动子元件分析
利用花生Tifrunner基因组及其注释文件,构建花生4CL基因家族成员的种内共线性图谱,将花生染色体信息文件、共线性文件、基因密度文件以及基因位置文件利用TBtools进行可视化。为了分析花生4CL基因启动子的顺式作用元件,从花生Tifrunner基因组数据中提取了每个Ah4CL基因编码序列(CDS)上游2 000 bp的启动子序列。使用PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行分析,并用TBtools将元件进行可视化。
1.2.5 花生4CL基因转录组数据分析
表达模式分析转录组数据来源于NCBI的SRA数据库中研究课题SRP302163,花生叶片ABA、干旱及盐处理转录组数据源于研究课题SRP215177,通过Cufflinks[23]分析FPKM(fragments per kilobase of transcript per million fragments)值,并用TBtools绘制热图。
1.2.6 花生组织RNA提取、反转录及RT-qPCR
用RNAiso Easy(TaKaRa)RNA提取试剂盒进行总RNA的提取,随后用PrimeScript™ FAST RT reagent Kit with gDNA Eraser(TaKaRa)反转录试剂盒进行cDNA第一链的合成,最后使用2 × SYBR Green qPCR Master Mix(Selleck)在LineGene9600荧光定量PCR仪上进行RT-qPCR,使用2-ΔΔCt方法计算检测基因的相对转录量[24],内参基因为花生Actin7(LOC112783163)基因。RT-qPCR所用引物见表1。
Table 1 Genetic primers for RT-qPCR
| 基因名称 Gene name | 正向引物 Forward primer (5′‒3′) | 反向引物 Reverse primer (5′‒3′) |
|---|---|---|
| Ahactin7 | TTGGAATGGGTCAGAAGGATGC | AGTGGTGCCTCAGTAAGAAGC |
| Ah4CL21 | GAGAGAGCGAGGATGAAGGC | AGCTCCTCTGACCACGATCT |
| Ah4CL22 | TCTTGGACAGAGCGGGGATA | GTGTCCTCTTCCAATGCCGA |
| Ah4CL28 | GCTACACTTGGACCCTTGCT | GGCAGAGGAAGGATGGTGTC |
| Ah4CL37 | AGCCCTTGTGCCTTTCTCTC | CATGGCCCTCAACTGAACCT |
| Ah4CL49 | CGAGAATTTGAGCAGCGTGG | ACTCCACCATCTCCTCCTCC |
| Ah4CL53 | CTCTGCACTCCCTCAACCTG | GCCTGGTTTACGCTCTCCTT |
2 结果
2.1 花生4CL基因家族鉴定及蛋白理化性质分析
借助NCBI中花生品种Tifrunner的全基因组数据,通过HMM文件(PF00501,PF13193)的筛选以及NCBI CDD和Pfam数据库的分析,最终共鉴定出56个Ah4CL基因(附表1)。为了便于后续分析,依据基因在染色体上的位置对所有基因进行系统的命名。利用ExPASy-ProtParam工具对Ah4CL基因家族蛋白的基本特性进行分析(附表1)。Ah4CLs编码多种蛋白质,氨基酸长度范围239‒1 208,蛋白的分子量范围为26.52‒134.62 kD,理论等电点范围为5.5‒9.22。脂肪族指数在80.2‒103.13之间,表明不同Ah4CL蛋白表现出不同程度的热稳定性。同时,不稳定性指数在25.51‒48.79之间,其中低于40的Ah4CL蛋白有39个,高于40的蛋白有17个,表明后一类Ah4CL蛋白质的稳定性较低。GRAVY值表示Ah4CL蛋白的疏水或亲水性特征,范围为-0.367‒0.139,其中有39个负值,表明这些蛋白为亲水性蛋白,剩余17个为正值,为疏水性蛋白。
2.2 花生4CL蛋白的进化分析
为了进一步分析花生4CL家族的系统发育以及功能特征,利用MEGA软件对花生和拟南芥的4CL蛋白进行进化分析(图1)。随后,根据已有报道所描述的4CL基因家族的分类方法对它们的聚类情况进行分类。所有4CL基因家族成员被划分为7个分支(Clade 4CL和A‒F)。位于Clade 4CL的花生4CL蛋白,共有11个。其中,花生4CL蛋白Ah4CL14、Ah4CL34、Ah4CL35、Ah4CL36和Ah4CL43与拟南芥中的At4CL3蛋白有较强的同源性。其余6个蛋白分别为Ah4CL3、Ah4CL4、Ah4CL10、Ah4CL29、Ah4CL30和Ah4CL38,与拟南芥中的At4CL1、At4CL2和At4CL4具有较高的同源性。这些蛋白聚类到Clade 4CL,表明它们可能参与木质素和类黄酮的生物合成。进化枝A‒E均包含拟南芥At4CL-like蛋白,其中,Ah4CL1和Ah4CL27聚集在Clade A中,Ah4CL8、Ah4CL23、Ah4CL33和Ah4CL50聚类在Clade B中,Ah4CL12、Ah4CL25、Ah4CL40和Ah4CL54聚类在Clade C中,Ah4CL6和Ah4CL31聚类在Clade D中,在Clade E中有Ah4CL24、Ah4CL26、Ah4CL51和Ah4CL56。在Clade F中,缺少At4CL-like蛋白,只有Ah4CL蛋白。基于之前的文献报道,该分支可能含有AAE(acyl activating enzymes)或AAEL(acyl activating enzyme-like)蛋白。这些4CL-like蛋白可能在植物代谢和多种天然化合物合成途径中发挥作用。
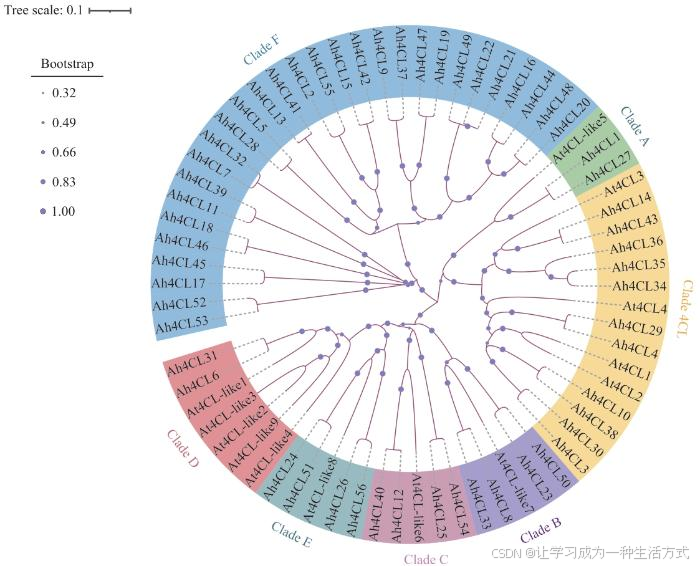
图1 花生与拟南芥4CL蛋白的系统进化分析
不同区域颜色表示不同分类亚组分支,包括Clade 4CL与Clade A‒F
2.3 花生4CL基因的染色体分布及共线性分析
通过分析花生4CL基因在染色体上的位置(图2),发现有56个4CL基因不均匀地分布在花生A和B基因组的20条染色体上。5和15号染色体Ah4CL基因密度最高,为10.71%,其次是1和13号染色体,Ah4CLs密度为8.93%。接着是7和11号染色体,Ah4CLs密度为7.14%。17和19号染色体,Ah4CLs基因密度为5.36%。2、3、4、8、12、14、18和20号染色体含有2个Ah4CL基因,基因密度为3.57%。含有1个Ah4CL基因的为6、9、10和16号染色体,基因密度为1.79%。
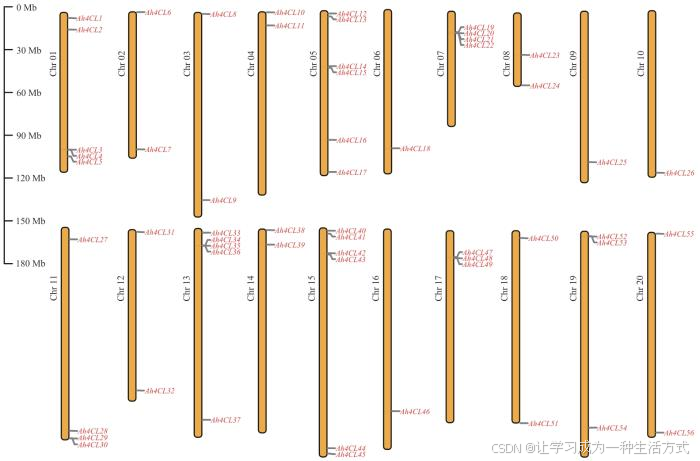
图2 花生4CL基因染色体分布分析
对花生4CL基因家族的56个基因进行种内共线性分析(图3),发现共线性基因有37对。在Clade 4CL分支中,Chr1上的Ah4CL3与Chr11上的Ah4CL29具有共线性,Chr4上的Ah4CL10与Chr14上的Ah4CL38基因共线,Chr5上的Ah4CL14分别与Chr13上的Ah4CL34和Chr15上的Ah4CL43存在共线性,另外,Chr13上的Ah4CL34和Chr15上的Ah4CL43之间也存在共线性。全基因组复制或多倍体化、串联复制以及片段复制事件在花生4CL基因家族的进化中起着至关重要的作用。
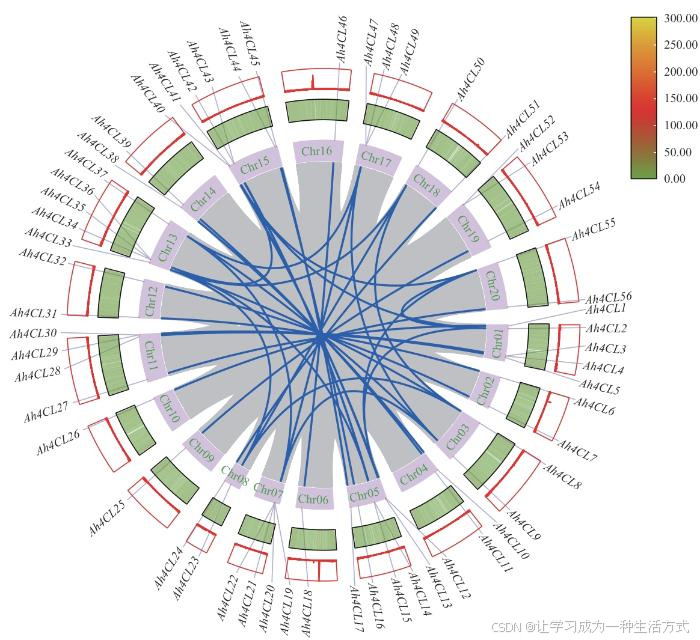
图3 花生4CL基因家族成员共线性分析
2.4 花生4CL蛋白保守基序、结构域以及基因结构分析
为了更深入地了解Ah4CL基因家族,对每个Ah4CL蛋白的保守基序、结构域和基因结构进行分析(图4)。利用MEME工具预测Ah4CL基因家族的保守蛋白基序,在Ah4CL基因家族中,鉴定了10个不同的基序(Motif 1‒10)(表2)。Ah4CL蛋白的保守基序数量在6‒10之间(图4-B)。在同一个聚类分支中,花生4CLs具有相似的保守基序组成,表明它们之间有密切的进化关系。Ah4CL基因家族的所有成员在NCBI CDD数据库中都具有4CL结构域。同时,在Pfam数据库中都具有AMP binding和AMP binding_C结构域(图4-C)。Ah4CL基因的外显子数量在1‒18个之间,在同一聚类分支的Ah4CL基因具有相似的内含子‒外显子分布结构(图4-D)。
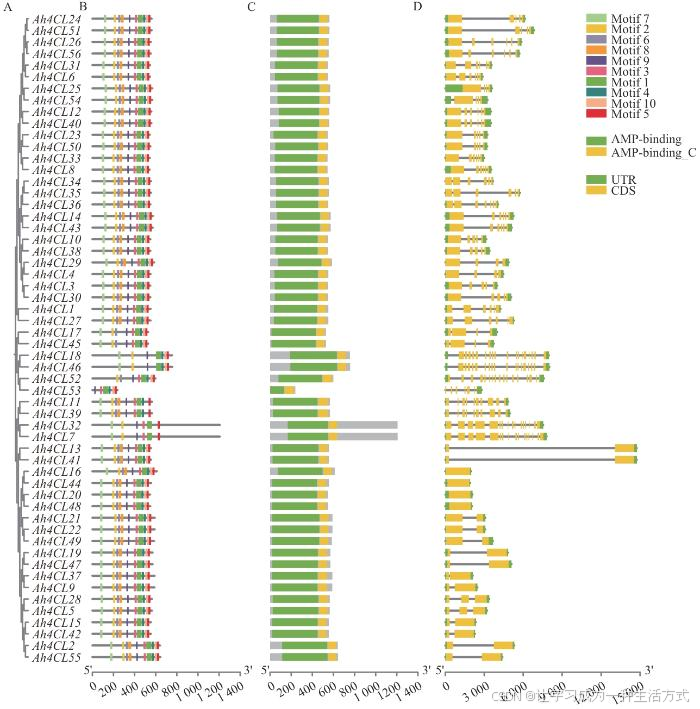
图4 花生4CL家族成员进化关系(A)、保守基序(B)、保守结构域(C)及基因结构(D)分析
Table 2 Conserved motif sequences of peanut 4CL family proteins
| 基序 Motif | 序列 Sequence | E值 E_value |
|---|---|---|
| Motif 1 | GWLHTGDLGYIDEDGYJFIVDRLKELIKYKGEQVAPAELEAVLYSHP | 3.8e-1 534 |
| Motif 2 | LLYSSGTTGLPKGVVLTHRGL | 1.8e-653 |
| Motif 3 | GEICIRGPTIMKGYLKBPEAT | 2.1e-581 |
| Motif 4 | DAAVVPRPDEEAGEVPCAFVV | 4.8e-540 |
| Motif 5 | VVFIDSJPKTSTGKILRKDLR | 2.8e-488 |
| Motif 6 | KSEDVYLWTLPMFHVNGLCFP | 2.8e-448 |
| Motif 7 | SPAFYELHLAVPMAGAVLTTANP | 7.2e-402 |
| Motif 8 | AVGGTNVCMRKFDAKAILEAIEKHKVT | 5.4e-457 |
| Motif 9 | PGAIVSQGYGMTETG | 6.9e-322 |
| Motif 10 | ITEEEIIEFCAKQVAPYKRPK | 3.5e-402 |
2.5 花生4CL基因启动子顺式作用元件分析
为了分析花生4CL基因家族成员所参与的潜在的转录调控机制,对每个成员上游2 000 bp启动子序列进行顺式作用元件预测(图5)。结果显示,花生4CL基因家族启动子区域富含多种顺式作用元件,包括光响应元件、非生物胁迫(低温、干旱等)响应元件、植物激素(脱落酸、茉莉酸甲酯、生长素、赤霉素和水杨酸)响应元件,以及生长发育(类黄酮生物合成和昼夜节律)响应元件。鉴定花生4CL基因家族成员中有很大一部分启动子区包含有脱落酸和干旱响应相关的顺式作用元件,表明Ah4CLs在非生物胁迫响应以及调控过程中可能发挥着关键的功能。
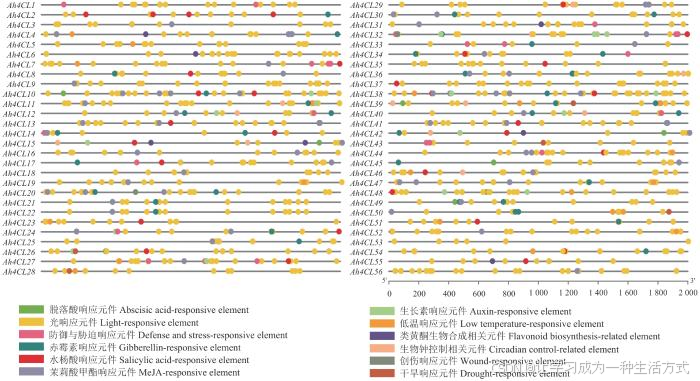
图5 花生4CL基因启动子区顺式作用元件分析
2.6 花生4CL基因组织表达模式分析
基因组织特异性表达模式探索可以帮助我们更好的分析Ah4CL基因潜在的生物学功能。Ah4CL基因家族不同成员具有不同的表达模式(图6-A),表明它们具有多方面功能。具体而言,Ah4CL15、Ah4CL42、Ah4CL29、Ah4CL38、Ah4CL5和Ah4CL10在根中表达水平较高,Ah4CL15、Ah4CL18、Ah4CL46和Ah4CL52在叶片中表达量相对较高,Ah4CL17、Ah4CL45、Ah4CL28和Ah4CL8在花中表现出更高的表达水平,可能在花生生殖生长过程中发挥着重要的功能。另外,Ah4CL15、Ah4CL42以及Ah4CL5在花生种子中有较高表达,可能在花生籽粒正常生长发育过程中起着重要的作用。Ah4CL基因的多样性以及不同组织特异表达模式保障着花生发育以及生理功能的正常运行。
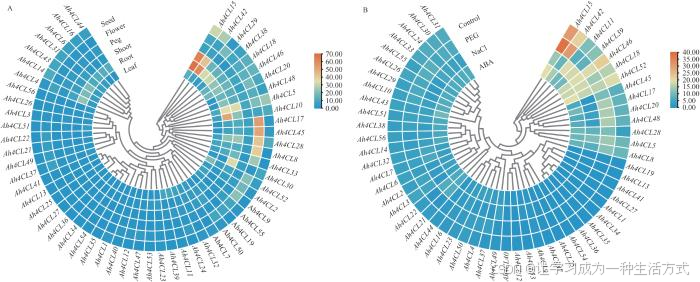
图6 花生4CL基因在不同组织(A)及在干旱、盐及ABA处理后(B)的表达分析
2.7 花生4CL基因在盐和干旱胁迫下的表达水平分析
为了探究Ah4CL基因在花生非生物胁迫应答中的潜在功能,首先通过转录组数据分析Ah4CL基因在盐、干旱胁迫以及ABA处理下的表达水平变化(图6-B)。结果显示,Ah4CL基因对盐和干旱胁迫的响应存在差异,表明不同的Ah4CL在非生物胁迫中发挥的作用不同。其中,Ah4CL28和Ah4CL37基因在模拟干旱(PEG)处理后表达量上调2倍以上。盐处理后,Ah4CL21、Ah4CL22、Ah4CL28、Ah4CL49和Ah4CL53的表达水平显著升高。ABA在植物非生物胁迫响应过程中发挥着重要的功能,ABA处理下Ah4CL21、Ah4CL22、Ah4CL28、Ah4CL38、Ah4CL43、Ah4CL49和Ah4CL53表达量也上调2倍以上。值得注意的是,Ah4CL21、Ah4CL22、Ah4CL28、Ah4CL49和Ah4CL53在盐和ABA处理后均出现上调,表明在花生中这些基因可能在ABA信号调节通路中参与盐胁迫调控,尤其是Ah4CL28在干旱、盐以及ABA处理下均出现上调,表明这一基因在花生应对非生物胁迫中可能发挥着至关重要的作用。
RT-qPCR实验结果显示,在PEG6000处理下,Ah4CL28的表达量在叶片中出现显著上调(图7-A),在根中仅在处理24 h时出现显著上调(图7-C),Ah4CL37的表达量在干旱处理6与12 h时,在叶片中有显著的上调(图7-B),而在根中仅在处理后24 h有显著增加(图7-D)。
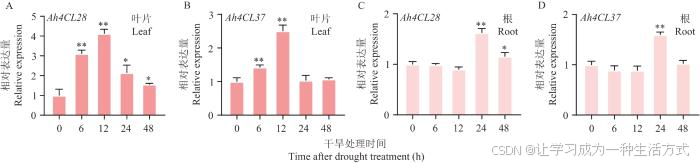
图7 模拟干旱处理后Ah4CL28及Ah4CL37表达水平变化
通过Student’s t检验进行显著性分析,*表示P<0.05,**表示P<0.01。下同
在盐处理12与24 h时,Ah4CL21的表达量在叶片中显著上调(图8-A),而在根中仅在处理后12 h有显著增加(图8-F);在盐处理12、24与48 h时,Ah4CL22的表达量在叶片中显著上调(图8-B),而在根中,在处理后12与24 h时表达量出现显著增加(图8-G);在盐处理6、12与24 h时,Ah4CL28的表达量在叶片中显著上调(图8-C),而在根中,在处理后6、12、24与48 h时有显著增加(图8-H);在盐处理12、24与48 h时,Ah4CL49的表达量在叶片中显著上调(图8-D),而在根中仅在处理后12 h时有显著增加(图8-I);在盐处理6、24与48 h时,Ah4CL53的表达量在叶片中显著上调(图8-E),而在根中,在处理后6、12与48 h时有显著增加(图8-J),进一步表明这些Ah4CL基因家族成员可能在花生应对干旱与盐胁迫中起着关键作用。
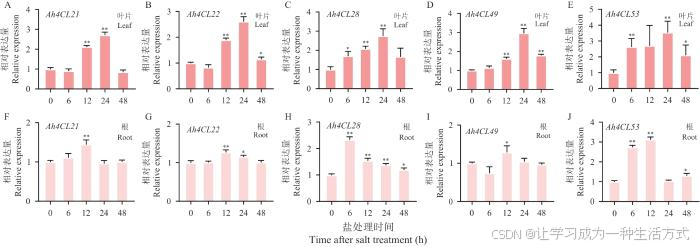
图8 盐处理后Ah4CL21、 Ah4CL22、 Ah4CL28、 Ah4CL49及Ah4CL53表达水平变化
3 讨论
3.1 花生Ah4CLs的鉴定与基本特性分析
苯丙烷类物质的合成是陆生植物正常生长发育及抵御外界复杂环境所必需的[25]。4CL作为苯丙烷类物质代谢过程中的关键酶发挥着重要的作用[26]。4CL酶活性影响着多种化合物积累,尤其是类黄酮和木质素[14]。在这项研究中,通过对花生品种Tifrunner进行全基因组分析,鉴定到花生中56个4CL基因成员。依据已有报道[11]的研究分类,将整个花生4CL基因成员系统地划分为7个不同的分支,分别记为Clade 4CL和A‒F。拟南芥中At4CL1‒4位于Clade 4CL分支,已有研究表明At4CL1‒4进一步分为2种类型,其中At4CL1、At4CL2和At4CL4属于I型,与木质素生物合成有关,而At4CL3属于II型,与木质素以外的苯丙烷类物质合成相关[27]。Ah4CL14、Ah4CL34、Ah4CL35、Ah4CL36和Ah4CL43与At4CL3位于同一个子分支,同源关系较近,表明这些Ah4CL蛋白可能参与木质素以外的苯丙烷类物质合成,如类黄酮。另一方面,Ah4CL3、Ah4CL4、Ah4CL10、Ah4CL29、Ah4CL30和Ah4CL38与At4CL1、At4CL2和At4CL4聚集在同一个子分支,这些蛋白可能与木质素合成相关。Clade A‒F中的Ah4CL蛋白与Clade 4CL蛋白都具有AMP-binding和AMP-binding _c结构域以及4CL结构域,进化关系较近,但是在功能上这些4CL-like蛋白可能会有差异。Ah4CLs在染色体上的分布不均匀,但是互相之间却有许多基因表现出高度的相似性和共线性。56个Ah4CL基因家族成员共线性基因有37对,如Ah4CL3与Ah4CL29、Ah4CL10与Ah4CL38和Ah4CL34与Ah4CL43等。多倍体化、串联复制以及片段复制事件可能在花生4CL基因进化中发挥着关键的功能。基因的结构及保守结构域的变化影响着基因的功能[28]。同一聚类分支的大多数Ah4CL基因表现出相似的内含子‒外显子结构,以及相似的保守基序的数量和分布,这些特征反映出Ah4CL基因在进化过程中的功能保守性。
3.2 花生Ah4CLs在干旱与盐胁迫中的响应分析
花生在大田生长过程中会遭遇到多种非生物胁迫,尤其是干旱与盐胁迫[29-30],已有的研究表明4CL基因可以响应干旱与盐胁迫,不同的4CL成员对于干旱与盐胁迫的响应程度也不同[15, 31-32]。花生4CL基因家族的鉴定与分析将有助于进一步探究花生面对干旱与盐胁迫的调控机理。顺式作用元件影响着基因表达调控[33],基因启动子上一系列不同的顺式作用元件可能与多种蛋白进行核酸蛋白互作,增加了基因调控的特异性及复杂性。已鉴定的多数Ah4CLs含有对脱落酸和干旱响应相关的顺式作用元件,而ABA在植物非生物胁迫的调控中发挥重要作用[34],表明Ah4CLs是花生应对非生物胁迫调控的关键基因。在花生ABA处理之后的转录组数据中发现,Ah4CL21、Ah4CL22、Ah4CL28、Ah4CL38、Ah4CL43、Ah4CL49和Ah4CL53表达水平上调2倍以上,表明这些Ah4CLs可能参与到ABA信号通路来调控花生的抗逆性。值得注意的是Ah4CL21、Ah4CL22、Ah4CL28、Ah4CL49和Ah4CL53的表达在盐处理后也出现显著的升高,进一步表明这5个Ah4CLs可能在ABA信号通路中来调控花生的抗盐性。此外,在模拟干旱处理下,Ah4CL28和Ah4CL37的表达出现显著上调,表明这2个Ah4CLs基因可能在干旱胁迫中起作用。有趣的是,Ah4CL28在ABA、盐以及干旱胁迫下均出现显著上调,表明这一基因在非生物胁迫中的重要性。4CL催化产生多种植物酚类次级产物,尤其是木质素与类黄酮,这些组分在植物抗逆性调控过程中发挥关键作用[35-36]。花生中Ah4CLs在木质素及类黄酮等代谢物合成及抗逆性调控中的功能仍需进一步通过遗传材料进行验证与分析。
4
4 结论
在花生中共鉴定出56个Ah4CLs,不同Ah4CLs之间存在差异,但相同聚类分支又具有保守性。多倍体化、串联复制以及片段复制事件在Ah4CLs基因进化中可能发挥关键作用。Ah4CLs响应多种发育、激素以及逆境胁迫信号。Ah4CL28和Ah4CL37可能参与花生干旱胁迫响应,而Ah4CL21、Ah4CL22、Ah4CL28、Ah4CL49和Ah4CL53可能参与花生盐胁迫响应过程。
