《Resolving tissue complexity by multimodal spatial omics modeling with MISO》
概念
多模态空间组学:简单来说,就是同时研究生物组织里的多种分子信息(比如基因表达、蛋白质、代谢物、表观遗传标记等),而且这些信息还带有空间位置。
MISO(MultI-modal Spatial Omics)是这篇论文提出的一种算法,专门用来分析这些复杂的多模态空间组学数据。它的目标是把不同类型的数据(比如基因表达和组织切片的显微镜图像)整合起来,找出组织里不同的功能区域(叫“空间域”),而且还能处理超大规模的数据,比如亚细胞级别的精细数据。
MISO算法的工作流程
MISO的工作流程(图1)包括以下关键步骤:
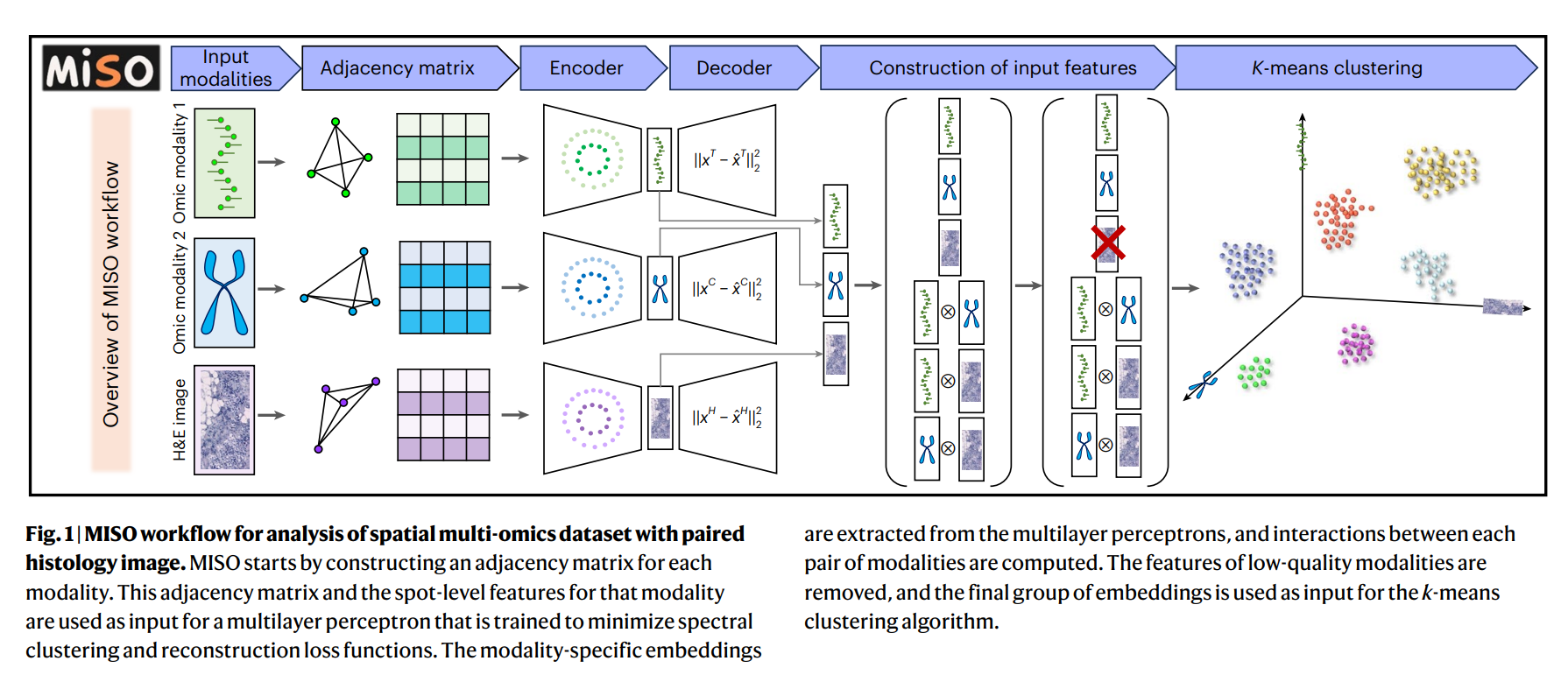
2.构建模态特异性邻接矩阵,捕捉数据点之间的相似性,利用神经网络(SpectralNet启 发)生成低维特征向量。
3.对于组织学成像(如H&E染色图像),使用预训练的层次视觉变换器(ViT)模型提取 局部和全局特征,生成每个点的576维嵌入。
对于组织学图像,MISO用一个预训练的视觉变换器(ViT)模型,分析图像的局部和全局特征。比如,它会看一个细胞附近的小区域(局部特征),同时也考虑整个组织切片的模式(全局特征),最后给每个数据点生成一个576维的图像特征向量。
特征提取(”精华提取“):
- 对每种模态使用特定的多层感知器(MLP(一种神经网络))提取低维嵌入,基于光谱聚类和重构损失函数进行训练。
“多层感知器”(MLP)的神经网络,把高维数据(比如几千个基因的表达量)压缩成低维的“特征向量”,有点像把一大堆信息提炼成一个简洁的“摘要”。
“光谱聚类”该方法,基于数据点之间的相似性(通过“邻接矩阵”表示),确保提取的特征能捕捉数据的内在结构。
跨模态特征融合(”混出新味道“):
- 计算模态对之间的外积(”外积“一种数学操作),生成交互特征矩阵,捕捉模态间的相互作用(如基因表达与表观遗传数据的调控关系)。
为什么还要计算模态对?因为光有单独的特征还不够,MISO还会看不同模态之间的“互动”。比如,基因表达和染色质开放性(ATAC-seq)可能一起决定某个细胞的功能。
2.通过主成分分析(PCA)将交互特征降维至32维,与模态特异性特征保持一致。然后和单独模态的特征拼接在一起,形成一个包含所有信息的“综合特征”。低质量模态过滤(”丢掉烂食材“):
- 用户需通过视觉检查(图像模态)或定量指标(如基因表达的UMI计数、代谢物信号的空间分布)识别低质量模态,并决定是否纳入聚类。
某些数据质量不好,比如图像有模糊或染色不均,基因数据测序深度不够,通过检查将低质量的特征剔除,只保留高质量的特征和它们的交互特征。
K均值聚类(”分菜,装盘“):
- 将模态特异性和交互特征向量拼接后,输入K均值聚类算法,生成反映生物学特性的空间域。每个域代表一个生物学上独特的功能区域,比如肿瘤区域、免疫细胞聚集区等。
大规模数据集优化:
- 对于大规模数据集(如MERFISH生成的数据),MISO通过稀疏邻接矩阵(基于k近邻,k=100)降低存储需求,提高计算效率。
MISO的计算效率突出,可在单GPU上处理中等规模数据集(1,000-10,000个点)在1分钟内完成,并能扩展至包含数十万点的超大规模数据集。
应用案例与结果
文章通过16个数据集(涵盖小鼠、人类、斑马鱼的多种组织和模态)展示了MISO的性能,以下是主要应用案例的总结:
膀胱癌10x Visium V2数据(利用istar工具识别微米级案例)(图2)
总结:MISO像个“显微镜”,能从复杂的数据里精准挖出细小的HEV结构,其他方法要么太“粗糙”,要么直接“看不见”。
- 数据集:转录组+组织学成像(H&E),使用iStar增强分辨率至4×4 µm²超像素。
- 目标:识别三级淋巴结构(TLS)中的高内皮小静脉(HEV)。
- 结果:MISO精确识别TLS1和TLS2中的HEV(误分非HEV超像素仅10个和6个),灵敏度和特异性均高于SpatialGlue(误分289和381个)和MUSE(无法生成HEV相关聚类)。
- 评估:通过类内相关系数(ICC)比较,MISO在RNA和组织学数据的聚类一致性上优于其他方法。
胃癌10x Xenium数据(数据庞大的案例)(图3a-e)
- 数据集:单细胞分辨率转录组(377个基因,696,314个细胞)+组织学成像。
- 目标:区分病理学家标注的组织结构(如低黏附性癌、侵袭性分散肿瘤细胞、淋巴聚集等)。
- 结果:MISO准确区分低黏附性癌(聚类14)和分散肿瘤细胞(聚类5),并识别小规模淋巴聚集,减少手动标注时间。MISO利用组织学特征成功区分表达相似标记基因(如ERBB2)的癌和黏膜区域。
- 优势:由于内存限制,MUSE和SpatialGlue无法处理此大规模数据集,而MISO展现了处理能力。
总结:MISO像个“超级大脑”,在数据量大、基因信息少的情况下,依然能精准找到复杂的组织结构,而其他方法直接“卡壳”了。
3.结直肠癌10x Visium HD数据(展示MISO处理亚细胞分辨率和全转录组数据的超强能力)(图3f-i)
1.数据集:亚细胞分辨率全转录组(18,085个基因,545,913个8×8 µm²区域)+组织学成像。
2.目标:区分肌肉、正常结肠黏膜和侵袭性癌。
3.结果:MISO识别侵袭性癌的多个子类,其中聚类4与SPP1+巨噬细胞共定位,提示更具侵袭性的肿瘤细胞(SPP1在聚类4中上调,log2倍变化1.06,P=1.22×10⁻⁵⁶)。
4.优势:MISO通过提取组织学特征,捕捉短程和远程依赖,优于MUSE和SpatialGlue(后者因内存限制无法分析)。
总结:MISO可以找到癌区,同时进一步将癌区分成几个子类,并与SPP1+巨噬细胞共定位,从而找到更侵袭性的癌细胞亚型。
4.小鼠胚胎(E13)空间ATAC-RNA-seq数据(涉及两种复杂的模态(转录组和表观遗传组))(图4)
1.数据集:转录组+染色质可及性+组织学成像,聚焦前脑端脑区域。
2.目标:区分端脑的脑室区(VZ)、底板层和皮质层。
3.结果:MISO唯一区分了VZ(SOX2高表达,聚类3)、底板层(GAD2高表达,聚类1)和皮质层(TBR1高表达,聚类6),验证了其整合复杂模态的能力。t-SNE分析显示MISO聚类在RNA和ATAC模态上分离度高,优于MUSE和SpatialGlue。
总结:MISO能整合RNA、ATAC和图像三种模态,捕捉复杂的空间和分子关系。
5.小鼠海马空间转录组与代谢组数据(支持三模态整合)(图5a-c)
- 数据集:转录组+代谢组(MALDI-MSI)+组织学成像,增强分辨率后对齐空间坐标。
- 目标:细分海马锥体层(CA1sp、CA2sp、CA3sp)。
- 结果:MISO准确区分CA1sp、CA2sp和CA3sp(包括CA3的三个子区域),而MUSE和SpatialGlue仅区分CA1sp和CA3sp。MERFISH数据验证了MISO在CA2sp/CA3sp边界的精确性。
总结:MISO像个“精密雕刻师”,能把海马的细微结构雕得清清楚楚,其他方法只能看到“大轮廓”。
MISO能整合三种模态(转录组、代谢组、图像),捕捉更细致的结构。
6.人类扁桃体空间基因与蛋白表达数据(展示了MISO处理三种模态(转录组、蛋白质组、组织图像)的能力)(图5d-h)
- 数据集:转录组+蛋白质组(35个蛋白)+组织学成像。
- 目标:定位生发中心。
- 结果:MISO在所有模态组合下的F1分数最高(0.90,三模态;0.86-0.88,双模态),优于MUSE(最高0.87)和SpatialGlue(最高0.83)。
总结:MISO能处理三种模态(转录组、蛋白质组、组织图像)的能力,尤其是在免疫组织中定位关键结构。MISO像个“免疫系统GPS”,能精准导航到生发中心这个“免疫大本营”,而其他方法可能会“迷路”。
其他应用(扩展数据图2-10,补充图2-5)
1. 小鼠前脑空间转录组数据(扩展数据图2)
- 数据集:10x Visium小鼠前脑(前部冠状切片),包含转录组和H&E图像。
- 目标:根据Allen Brain Atlas的标注,区分前脑的皮质层和其他结构。
- MISO的表现:
- MISO的聚类结果与Allen Brain Atlas的皮质层标注高度一致,准确区分了不同脑区(图2b)。
- 它的ICC(类内相关系数)在RNA和图像模态上都高于MUSE和SpatialGlue(图2c),说明聚类更一致。
- 即使图像有伪影(图2e),MISO依然通过RNA和交互特征保持了高精度。
- 对比:SpatialGlue对模态权重敏感,调整权重后结果不稳定(图2b);MUSE受图像伪影影响,聚类质量较低。
- 意义:MISO像个“脑区规划师”,能在图像质量不完美时,依然精准划分大脑区域,对研究脑功能和疾病(如癫痫)很有用。
2. 人类乳腺癌空间转录组数据(扩展数据图3)
- 数据集:10x Visium人类乳腺癌数据,包含转录组和H&E图像,病理学家标注了原位癌(DCIS)、侵袭性癌和脂肪区域。
- 目标:区分这些组织区域,找出侵袭性癌的子类。
- MISO的表现:
- MISO的聚类与病理学标注高度吻合(图3b),甚至在侵袭性癌区域找到一个子类,UMI计数明显低于其他子类(图3e),可能反映癌细胞的异质性。
- t-SNE图(图3d)显示MISO的聚类与基因表达模式一致,ICC也高于MUSE和SpatialGlue(图3c)。
- SpatialGlue对图像权重敏感,调整权重后仍无法识别脂肪区域(图3f)。
- 意义:MISO像个“癌症地图绘制者”,能找到癌细胞的细微差异,帮医生判断肿瘤的侵袭性,指导治疗。
3. 斑马鱼黑色素瘤空间转录组数据(扩展数据图4)
- 数据集:10x Visium斑马鱼黑色素瘤数据,包含转录组和H&E图像,但图像有模糊伪影(图4a)。
- 目标:在图像质量低的情况下,区分肿瘤和周围组织。
- MISO的表现:
- MISO识别出图像质量低,剔除了图像特异性特征,只用RNA和RNA×图像交互特征,依然得到高质量聚类(图4b)。
- MUSE受模糊伪影影响,聚类被图像质量“带偏”;SpatialGlue表现稍好但仍不如MISO(图4c)。
- 意义:MISO像个“抗干扰侦探”,即使图像模糊,也能从基因数据里挖出真相,适合处理实验中常见的数据质量问题。
4. 小鼠嗅球空间转录组数据(扩展数据图5)
- 数据集:10x Visium小鼠嗅球数据,包含转录组和H&E图像,标注了嗅球的层次结构。
- 目标:区分嗅球的各层(如颗粒细胞层、外丛状层)。
- MISO的表现:
- MISO的聚类与嗅球层次标注完美匹配(图5a-b),ICC高于MUSE和SpatialGlue(图5c)。
- MUSE把嗅球分成上下两部分,没能细化层次;SpatialGlue稍好但仍不如MISO。
- 意义:MISO像个“嗅觉解剖师”,能精准划分嗅球的结构,对研究嗅觉处理和神经退行性疾病(如帕金森)有帮助。
5. 人类乳腺癌空间基因与蛋白表达数据(扩展数据图8)
- 数据集:10x Visium人类乳腺癌数据,包含转录组、蛋白质组(3通道免疫荧光:DAPI、Vimentin、PCNA)和H&E图像。
- 目标:整合三种模态,区分组织结构。
- MISO的表现:
- MISO在所有模态组合(RNA+图像、RNA+蛋白、蛋白+图像、或三者一起)下都得到高质量聚类(图8a-d)。
- ICC显示MISO在RNA、蛋白和图像模态上的聚类一致性最高(图8f),比MUSE和SpatialGlue强。
- 意义:MISO像个“多才多艺的艺术家”,能用多种“颜料”(模态)画出组织的细致图像,适合研究复杂癌症微环境。
6. 小鼠脑空间转录组与代谢组数据(扩展数据图9-10,补充图5)
- 数据集:10x Visium和MALDI-MSI生成的小鼠脑冠状切片数据,包含转录组、代谢组和H&E图像。RNA质量低(图9e),数据量大(扩展数据图10用299,350个16×16像素伪点)。
- 目标:区分脑区(如海马、皮质),处理低质量RNA和大数据。
- MISO的表现:
- MISO剔除了低质量RNA特异性特征,只用RNA×代谢组和RNA×图像交互特征,依然准确区分脑区(图9a-d,图10a-d)。
- 在超大数据集(299,350个伪点)上,只有MISO能运行,MUSE和SpatialGlue因内存限制失败(图10f)。
- 意义:MISO像个“大数据处理机”,能在数据质量差、量大的情况下,依然挖出脑区的生物学结构,适合高通量组学研究。
7. 小鼠冠状脑空间CUT&Tag-RNA-seq数据(补充图3-4)
- 数据集:包含转录组和表观遗传标记(H3K27AC、H3K27ME3)的CUT&Tag数据,以及H&E图像。
- 目标:区分脑区,验证MISO在表观遗传数据上的表现。
- MISO的表现:
- MISO准确区分了脑区的功能区域,ICC高于MUSE和SpatialGlue,展现了整合表观遗传和转录组的能力。
- 意义:MISO像个“表观遗传解码器”,能揭示基因调控和表达的空间关系,对研究脑发育和疾病(如癫痫)有帮助。
MISO的优点与局限性
优点:
- 多模态整合:支持任意数量的模态(转录组、蛋白质组、表观遗传组、代谢组、组织学成像等)。
- 计算效率:快速处理中小规模数据集,稀疏矩阵优化支持大规模数据。MISO能在1分钟内处理1,000-10,000个点的数据集,用稀疏矩阵还能搞定几十万个点的大数据集。例如胃癌的那个Xenium数据集(69.6万个细胞)
- 鲁棒性:通过过滤低质量模态和最小化超参数调整,适应真实世界中的数据挑战。MISO能处理低质量数据,比如模糊的图像或低UMI计数的基因数据。它让用户决定是否剔除低质量模态,还通过交互特征保留部分信息。
- 高精度:在多种生物学结构识别中优于MUSE和SpatialGlue,尤其在细粒度结构(如HEV、CA2sp)上表现突出。
局限性:
- 图像模态限制(仅H&E):目前仅支持H&E染色图像的特征提取,不适用于其他成像类型(如三色法、免疫荧光、DAPI)。
- 无监督性:需用户解释聚类结果与生物学结构的关联。比如“这个聚类对应的是肿瘤还是正常组织?”这可能需要病理学家的专业知识。
- 模态贡献评估:无法直接量化各模态对聚类的贡献或自动检测低质量模态,需用户手动评估。
跟其他方法的对比
- MUSE:擅长转录组+图像,但不支持代谢组或蛋白质组,内存需求高,处理大数据时容易“翻车”。
- SpatialGlue:能处理两个组学模态,但不支持图像,超参数调整麻烦,对低质量模态敏感。
- MISO:综合能力最强,支持多模态,超参数少,内存优化好,适合大数据和复杂场景。
MISO如何处理H&E图像特征
H&E图像(组织学切片染色图像)是空间组学的重要模态,提供了组织的空间结构信息。MISO的图像特征提取过程很聪明,像是把一张复杂的“组织画”拆解成数字化的“特征拼图”。具体步骤如下(论文第10页,补充图6有详细示意图):
- 用层次视觉变换器(ViT)模型:
- MISO用了一个预训练的‘’Hierarchical Image Pyramid Transformer (HIPT)‘’模型,训练过10,000多张全景切片图像,能捕捉组织的局部和全局特征。
- 比喻:这就像一个“超级画师”,既能看清画上的小细节(细胞形态),也能把握整幅画的布局(组织结构)。
- 分层提取特征:
- 局部特征:HIPT把大图像切成256×256像素的小块,每个小块用一个局部ViT模型处理,生成一个384维的“补丁嵌入”(patch embedding),描述小块的细节。
- 全局特征:把这些256×256小块的特征(CLS token,192维)输入一个全局ViT模型,分析它们在更大区域(4096×4096像素)里的关系,生成捕捉远程依赖的特征。
- 每个16×16像素的小补丁最终得到一个576维嵌入(384维局部+192维全局)。
- 生成点级特征:
空间组学数据点(比如Visium的55微米点或Xenium的单细胞)通常覆盖多个16×16像素补丁。MISO把一个数据点覆盖的所有补丁的576维嵌入取平均,生成这个点的图像特征。
这就像把一张画上的每个区域(数据点)用“颜色、纹理、结构”的综合描述概括出来。
为什么这很重要?
- 在结直肠癌案例中,MISO用图像特征捕捉癌细胞和SPP1+巨噬细胞的空间关系,区分了更侵袭性的癌区。
- 在扁桃体案例中,图像特征帮MISO识别生发中心的组织学特征(比如PCNA高表达区的结构),提高了F1分数。
- 这种局部+全局的特征提取让MISO能看到组织里的“微观细节”和“宏观格局”,比只看基因数据的MUSE或SpatialGlue更全面。
