从0开始学习R语言--Day55--弹性网络
通常来说,样本数据的数据个数会远大于特征数,但是当我们遇到特殊数据,比如基因数据,可能会有成百上千甚至上万地特征量,而样本个数只有几十个,此时如果直接做回归,由于特征数量很多,且有很多特征共线性较高,很容易过拟合,而能处理共线性的方法,又无法将特征的系数压缩为0,这样计算量会大大增加。
用弹性网络建模,其与其他不同的是,有两个惩罚项,L1负责控制特征系数(可以为0),做初步的筛选;L2负责剔除相关性高的特征,进一步减少计算量。
以下是一个例子:
library(glmnet)set.seed(123)
# 生成100个样本,20个特征(其中5个真实相关)
n <- 100
p <- 20
X <- matrix(rnorm(n * p), n, p)
# 真实系数:前5个非零,后15个为零
true_beta <- c(rep(2, 5), rep(0, p - 5))
# 生成响应变量(含噪声)
y <- X %*% true_beta + rnorm(n, sd = 0.5)# 设置alpha:0.5表示L1和L2各占一半(可调整)
# lambda通过交叉验证选择
fit <- cv.glmnet(X, y, alpha = 0.5, nfolds = 10)# 查看最优lambda
print(fit$lambda.min)# 系数(非零的即被选中)
coef(fit, s = "lambda.min")# 系数路径图
plot(fit$glmnet.fit, xvar = "lambda")
abline(v = log(fit$lambda.min), col = "red", lty = 2)# 交叉验证误差
plot(fit)# 生成新测试数据
X_new <- matrix(rnorm(10 * p), 10, p)
y_pred <- predict(fit, newx = X_new, s = "lambda.min")
输出:
[1] 0.0307822
21 x 1 sparse Matrix of class "dgCMatrix"s1
(Intercept) -0.0633934906
V1 1.9180107173
V2 1.9416672262
V3 2.0525999367
V4 1.9649569228
V5 1.9739324612
V6 .
V7 .
V8 -0.0422258534
V9 -0.0028998487
V10 0.0017927761
V11 0.0685678920
V12 .
V13 -0.0422331782
V14 0.0111743085
V15 0.0163985377
V16 .
V17 -0.0079250381
V18 .
V19 0.0008252632
V20 -0.0450508462
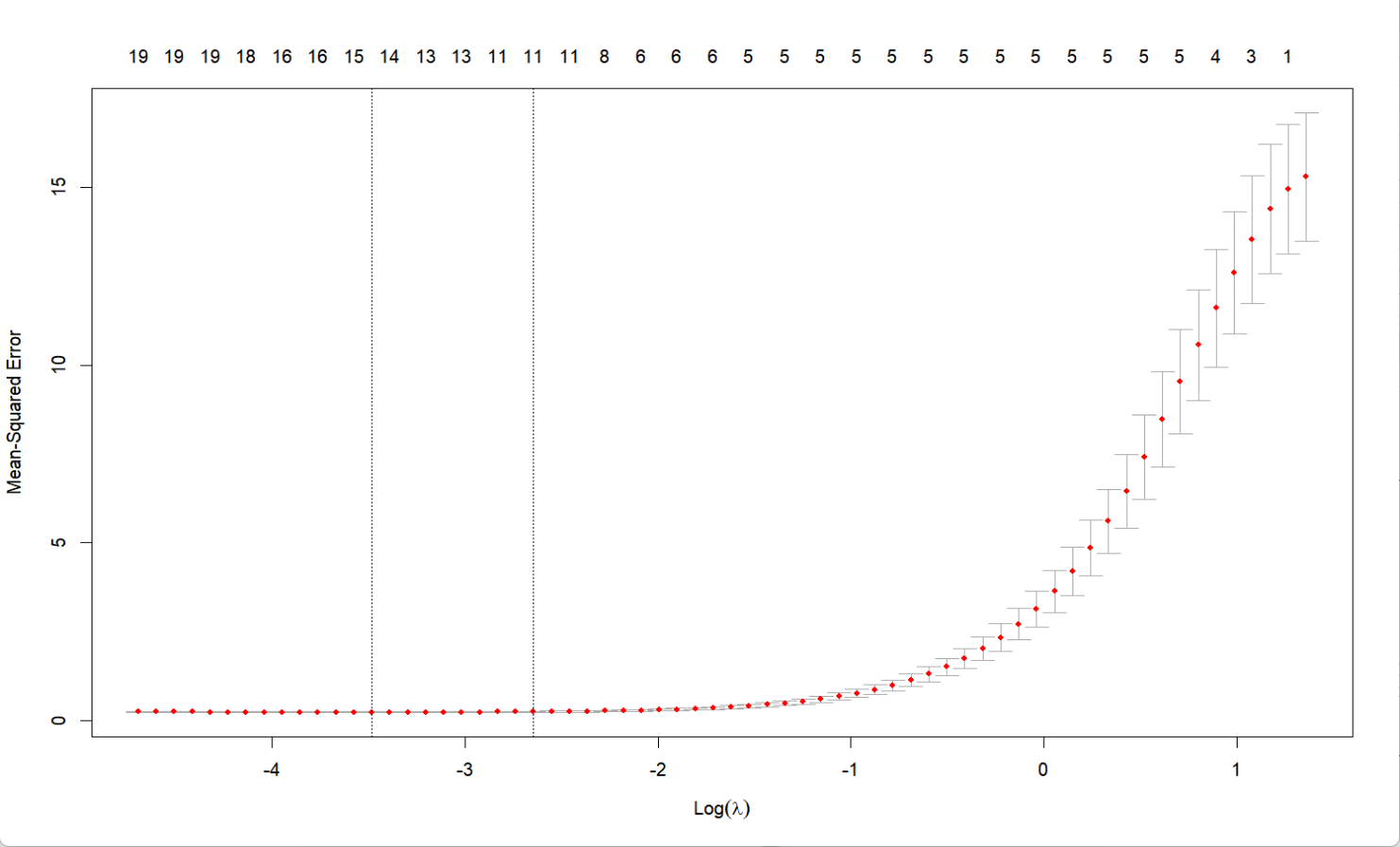
输出中可以看到,V1到V5对模型的影响比较大被保留,同时弱相关的特征也被保留了下来;而lambda=0.037可以最大程度的保留特征数量的同时平衡拟合优度。
