【数据分析】调控网络分析:调节因子在肿瘤样本中的表达相关性与生存效应分析
禁止商业或二改转载,仅供自学使用,侵权必究,如需截取部分内容请后台联系作者!
文章目录
-
- 介绍
-
- 数据准备与模拟
- 相关性分析与边表生成
- 网络可视化
- 结果展示与讨论
- 加载R包
- 模拟数据
- Spearman 相关 -> 边表
- 画图
- 所有代码
- 总结
- 系统信息
介绍
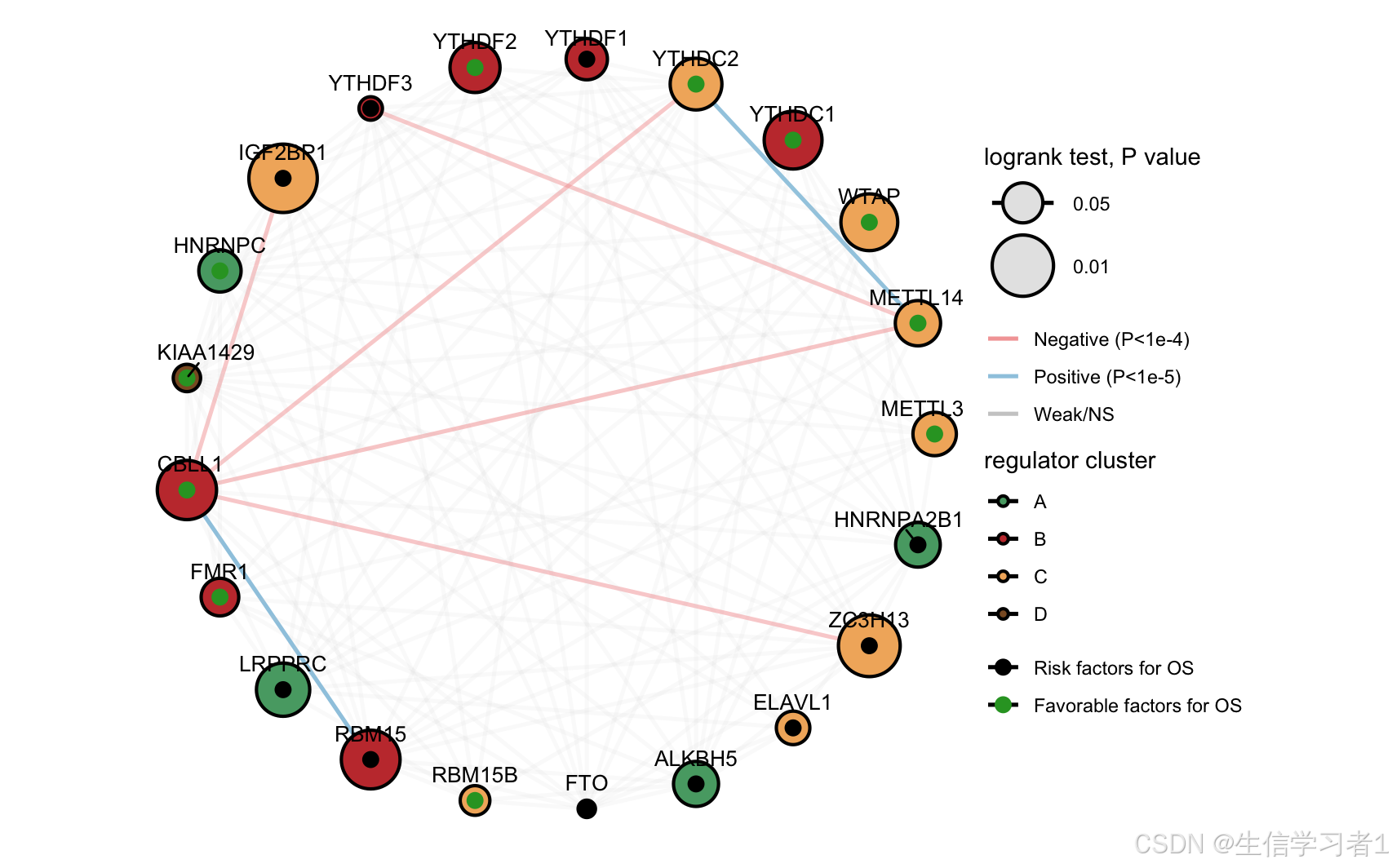
在生物医学研究中,N⁶-甲基腺苷(m⁶A)修饰作为一种重要的表观遗传调控机制,近年来受到了广泛关注。m⁶A 调控因子在多种生物学过程中发挥着关键作用,包括基因表达调控、细胞分化、免疫反应以及肿瘤发生等。为了深入理解这些调控因子之间的相互作用及其对细胞功能的影响,构建一个系统化的调控网络显得尤为重要。本研究通过模拟数据,展示了一个完整的分析流程,从基因表达矩阵的生成到基于 Spearman 相关性的网络可视化,旨在为研究者提供一个可复现的分析模板。
数据准备与模拟
研究首先定义了 21 个 m⁶A 调控因子,包括 METTL3、METTL14、WTAP 等,这些因子被随机分配到四个功能簇(A、B、C、D)中。每个基因还被赋予了生存效应(风险或有利)和 log-rank P 值,用于后续的节点属性展示。为了模拟基因表达数据,研究生成了一个包含 120 个样本的表达矩阵。通过潜在因子模型,确保了簇内基因表达的强正相关性和簇间基因表达的轻微负相关性,同时加入了随机噪声以模拟真实数据的复杂性。
相关性分析与边表生成
利用 Hmisc 包中的 rcorr 函数,研究计算了基因表达矩阵的 Spearma
