质谱数据分析环节体系整理
定性与定量
请分析质谱数据分析中的这几个概念,“特征离子识别,质谱图解析,分子式推断,同位素模式分析,结构推断,谱谱库匹配,匹配结果验证(如FEDR)”,并将其合并或分类或归类,使之成为一个方法体系,并作解释说明,
以下将质谱数据分析中的七个概念整合为分层递进的方法体系,按逻辑关系分为四个核心阶段,并解释各阶段的功能、关联及技术要点:
质谱数据分析方法体系
阶段一:数据预处理与特征提取
| 概念 | 功能说明 | 关键技术 |
|---|---|---|
| 特征离子识别 | 从原始谱图中提取关键离子信号(母离子、碎片离子、加合离子),过滤噪声,确定质荷比及强度。 | 峰检测算法(如Savitzky-Golay)、本底扣除、信噪比阈值过滤 |
阶段二:分子级信息推断
| 概念 | 功能说明 | 关键技术 |
|---|---|---|
| 同位素模式分析 | 利用同位素峰簇(如M+1, M+2峰)推断元素组成(如C、Cl、Br含量),辅助分子式验证。 | 同位素分布模型(如Beynon表)、高分辨率m/z匹配(误差<1 ppm) |
| 分子式推断 | 结合精确质量、同位素模式、元素组成规则(如氮规则),生成候选分子式集合。 | 穷举搜索、约束优化(如元素组成限制)、组合数学算法 |
阶段三:结构解析与候选生成
| 概念 | 功能说明 | 关键技术 |
|---|---|---|
| 质谱图解析 | 解读碎片离子断裂规律,推断官能团、骨架结构(如酯基断裂生成m/z 61)。 | 经典裂解规则库(如McLafferty重排)、中性丢失分析 |
| 结构推断 | 综合分子式、碎片信息、谱库匹配结果,生成候选化学结构。 | 图论算法(如结构生成树)、机器学习(如深度学习生成模型) |
| 谱库匹配 | 将实验谱图与标准谱库(如NIST、MassBank)比对,返回相似度最高的已知化合物。 | 相似度算法(如余弦相似度、概率匹配)、谱图对齐算法 |
阶段四:结果验证与优化
| 概念 | 功能说明 | 关键技术 |
|---|---|---|
| 匹配结果验证 | 通过独立方法(如保留时间、二级谱图一致性)验证候选结构的可靠性,减少假阳性。 | FEDR(False Discovery Rate)、交叉验证、多谱图一致性检验 |
体系逻辑关系与流程
各阶段技术关联与协同
- 特征离子识别 → 分子式推断
- 精确的母离子m/z(如300.1492 Da)是分子式推断的基础,同位素模式(如M+1峰强度)进一步约束元素组成(如碳原子数)。
- 同位素模式分析 → 结构推断
- 示例:检测到M+2峰强度为母离子的33% → 推断含1个Cl原子(³⁵Cl/³⁷Cl天然丰度比3:1),排除含Br的候选。
- 质谱图解析 + 谱库匹配 → 结构推断
- 若碎片离子m/z 91(苯基鎓离子)+ 谱库匹配到甲苯 → 推测结构含甲苯基团。
- 匹配结果验证 → 结果可靠性
- 通过FEDR控制假发现率:若谱库匹配得分>800,但保留时间偏差>0.5 min → 标记为低置信度结果。
体系优势与局限性
| 优势 | 局限性 |
|---|---|
| 分层递进:从数据到结构逐步聚焦,降低计算复杂度。 | 依赖预计算:谱库匹配需庞大的参考数据库,未知物可能无匹配。 |
| 多源证据融合:结合同位素、碎片、谱库等多维信息,提高推断准确性。 | 规则局限性:经典裂解规则不适用于非典型碎裂(如生物大分子)。 |
| 可验证性:FEDR等验证机制量化结果可靠性。 | 同分异构体挑战:相同分子式/碎片谱的不同结构(如对二甲苯/邻二甲苯)难以区分。 |
典型应用场景
- 天然产物鉴定
- 高分辨率质谱获取精确m/z → 同位素模式推断分子式 → 谱库匹配已知类似物 → 碎片解析官能团 → 验证UV光谱一致性。
- 环境污染物筛查
- 非靶向分析提取特征离子 → 分子式生成候选 → 谱库匹配农药数据库 → 结构碎片验证 → FEDR控制误报率。
- 代谢组学
- LC-MS/MS数据 → 特征离子提取 → 同位素模式区分同位素标记物 → 谱图树解析代谢通路 → 多样本验证生物学意义。
总结
该体系以**“数据→特征→分子→结构→验证”为核心逻辑,通过同位素模式分析和质谱图解析实现分子式与结构的双向约束,借助谱库匹配加速已知物鉴定,最终以FEDR量化结果置信度。其核心价值在于多模态证据融合与分层验证机制**,适用于从简单小分子到复杂生物样本的系统性结构解析。
以上内容由AI生成,仅供参考和借鉴
好的,这是一个非常核心且重要的问题。您列出的这些概念正是现代非靶向质谱分析,尤其是代谢组学、脂质组学等领域的核心流程。
将这些概念进行分类、整合,可以形成一个从原始数据到生物学洞察的、逻辑严密的方法体系。这个体系可以划分为 “数据预处理与特征提取”、“分子水平推断” 和 “验证与报告” 三大层级。
下图清晰地展示了这个完整的质谱数据分析方法体系,它遵循着从数据到知识的递进流程:
体系化解释说明
层级一:数据预处理与特征提取 (Data Preprocessing & Feature Extraction)
这是整个分析流程的基石,目标是将从仪器获得的原始信号转化为可信的、可供解释的化学实体(特征)。
-
1. 特征离子识别 (Feature Ion Identification):
- 内容: 从总离子流色谱图(TIC)中自动识别出色谱峰,并对其进行积分,得到每个峰的保留时间(RT)、m/z和强度(峰面积或峰高)。同时进行峰对齐,确保同一个特征在不同样本中能被正确匹配。
- 目标: 得到一个包含所有检测到的离子特征的矩阵(样本 × 特征)。
- 工具: XCMS, MZmine, MS-DIAL等。
-
2. 同位素模式分析 (Isotope Pattern Analysis):
- 内容: 对上一个步骤得到的每个特征,分析其同位素峰簇(如M, M+1, M+2峰的强度和间距)。这一步通常与“特征离子识别”中的峰解卷积(Peak Deconvolution) 紧密结合,用于区分加合离子、二聚体以及来自不同化合物的重叠峰。
- 目标: 识别出属于同一个分子的不同离子形式(如 [M+H]⁺, [M+Na]⁺, [M+NH₄]⁺),并将它们归并为一个化合物特征,从而减少数据冗余,提高后续定量的准确性。
- 工具: 内置于XCMS, MZmine等软件的算法。
关系: 这两个步骤紧密相连,同位素模式分析是特征离子识别过程中进行高级峰过滤和归并的关键依据。
层级二:分子水平推断 (Molecular-Level Inference)
在得到干净的化合物特征列表后,下一步就是推断这些特征的化学身份。这是整个流程的核心推理环节。
-
3. 分子式推断 (Molecular Formula Determination):
- 内容: 利用高分辨质谱提供的精确质量和同位素分布信息,计算出一个或多个可能的基础分子式(如 C₁₁H₁₇NO₅)。精确质量约束了元素组成的总质量,而同位素分布(特别是M+1/M+2的强度比)进一步约束了C, H, N, S, O等元素的可能数量组合。
- 目标: 为每个特征分配一个或一组最可能的元素组成。
- 工具: Sirius, Thermo Fisher Compound Discoverer中的元素组成分析模块。
-
4. 结构推断 (Structure Elucidation) & 4.1 谱图解析 (Fragmentation Analysis) & 4.2 谱谱库匹配 (Spectral Matching):
- 内容: 这是确定分子具体结构的步骤,通常需要MS/MS碎片离子数据。它包含两个相辅相成的子步骤:
- 4.1 谱图解析 (Fragmentation Analysis): 从机理上理解碎片离子是如何形成的,通过解读碎片来推导或验证子结构、官能团。这需要深厚的化学知识,通常由软件算法(如CSI:FingerID)或专家完成。
- 4.2 谱谱库匹配 (Spectral Matching): 将实验获得的MS/MS谱图与已知标准品的参考谱图库(如NIST, MassBank, GNPS)进行比对,通过相似度打分(如余弦相似度)来寻找最匹配的化合物。
- 目标: 由分子式推断走向确切的结构鉴定。
- 关系: 这是“同一个硬币的两面”。
- 谱谱库匹配是一种经验性、快速的方法,但严重依赖于数据库的覆盖度。
- 谱图解析是一种推理性、根本性的方法,可以鉴定新化合物,但对算法和专业知识要求极高。
- 工具: Sirius/CSI:FingerID (计算推理), MS-Finder, GNPS (库匹配), NIST MS Search。
- 内容: 这是确定分子具体结构的步骤,通常需要MS/MS碎片离子数据。它包含两个相辅相成的子步骤:
关系: 分子式推断是结构推断的必要前提。一个确定的分子式极大地缩小了候选结构的范围。随后,谱图解析和谱谱库匹配两种方法协同工作,共同指向最可能的结构。
层级三:验证与报告 (Validation & Reporting)
得到候选化合物后,必须对其可信度进行评估,并控制整体错误率。
- 5. 匹配结果验证 (Validation - e.g., FDR):
- 内容: 尤其是在大规模筛选中(如代谢组学),会产生大量鉴定结果,其中必然存在假阳性。需要采用统计方法对结果进行验证。错误发现率(FDR) 控制是最重要的方法之一。其核心思想是:同时搜索真实数据库和一个“诱饵”数据库(由不可能存在的分子式或谱图组成),通过计算诱饵库中的命中率来估计真实结果中的假阳性率。
- 目标: 评估整个鉴定结果集的可靠性,并给出每个鉴定结果的置信度水平(如Level 1-5注释)。
- 工具: 内置于GNPS, Compound Discoverer等流程中的FDR计算模块。
关系: 该步骤是确保最终报告结果科学严谨性的质量闸门。它是对前面所有推断步骤的最终审判。
总结与归纳
| 分析阶段 | 核心问题 | 所需数据 | 主要技术与概念 | 输出 |
|---|---|---|---|---|
| 1. 数据预处理与特征提取 | “有哪些信号?哪些信号属于同一个化合物?” | MS1 | 特征离子识别、同位素模式分析、峰解卷积 | 化合物特征表(m/z, RT, 强度) |
| 2. 分子水平推断 | “这个化合物是什么?” | MS1 (精确质量+同位素) MS/MS (碎片) | 分子式推断、谱图解析、谱谱库匹配 | 候选分子式、候选结构、相似度得分 |
| 3. 验证与报告 | “这些鉴定结果可信吗?” | 所有结果 | 匹配结果验证(FDR)、置信度分级 | 带置信度的最终注释列表(如Level 2) |
这个体系化的流程清晰地展示了现代计算质谱学如何像侦探破案一样,从海量的、嘈杂的原始信号(线索)出发,通过层层筛选、推理和验证,最终锁定目标的化学身份,从而为生物学和化学研究提供可靠的数据基础。
全部
请分析质谱数据分析中的这些概念,“解卷积、峰检测、峰对齐、同位素峰聚类、峰分组、缺失峰检测、峰补齐、峰特征矩阵生成、峰归一化、峰积分、峰标识、特征离子识别,质谱图解析,分子式推断,同位素模式分析,结构推断,谱谱库匹配,匹配结果验证(如FEDR),数据归一化,数据变换,非靶向定量,外标曲线校准、内标曲线校准、标准加入曲线校准,同位素标记曲线校准,批量效应校正,单变量统计分析,多变量无监督学习,多变量有监督学习,生物通路富集分析,机器学习特征筛选,机器学习模型,网络关联分析”,并将其合并或分类或归类,使之成为质谱数据分析的完整清晰的方法体系,并作解释说明,
嗯,用户这次要求整合一个庞大的质谱数据分析概念列表,看来是希望构建系统性的方法论框架。用户可能是质谱领域的研究者或学生,正在梳理知识体系,需要从零散概念中建立逻辑脉络。之前对话中用户对算法细节和分类逻辑表现出较高专业度,这次的需求明显更宏观,需要分层整合近40个概念。从概念列表看,用户覆盖了从原始数据预处理到生物意义挖掘的全流程。难点在于避免简单堆砌,而要体现技术间的递进关系。比如“峰检测”和“峰对齐”属于基础处理,“分子式推断”和“结构推断”属于结构解析,“通路富集”则是下游生物学分析,需要分层呈现。特别要注意几个易混淆点:同位素模式分析既是分子式推断的输入,又是独立分析步骤;校准方法中的内标/外标/标准加入需要明确应用场景差异;机器学习部分要区分特征筛选、模型构建和结果验证的层级关系。用户可能隐含的需求是:1)理解各概念在分析流程中的位置 2)掌握技术选择逻辑 3)建立完整工作流认知。因此回复要突出“数据流”主线,用箭头连接技术节点,并强调关键决策点(如何时用无监督学习、何时用有监督学习)。在结构设计上,采用“预处理→分子推断→结构解析→定量分析→统计建模→生物学解读”的六层架构比较合理。每层需说明输入输出和核心技术,比如定量分析层要区分校准方法的选择依据(外标适合简单样本,标准加入适合基质复杂样本)。最后补充技术选择决策树,帮助用户根据实际需求跳转阅读。
以下将质谱数据分析中的44个概念整合为分层递进的方法体系,按数据流程和功能逻辑划分为6大阶段,并解释各阶段的核心技术、关联逻辑及典型应用场景:
质谱数据分析完整方法体系
阶段一:原始数据预处理
| 概念 | 功能说明 | 核心技术 |
|---|---|---|
| 解卷积 | 分离重叠峰,提取真实离子信号(如LC-MS共流出峰) | 算法:Amplitude Deconvolution, Mass Spec Studio, XCMS |
| 峰检测 | 识别谱图中的真实峰(m/z-强度对),过滤噪声 | 算法:CentWave, MZmine, ChromaTOF |
| 峰对齐 | 校准不同样本/批次间的保留时间偏移(如LC-MS数据) | 算法:LOESS, Dynamic Time Warping (DTW), OpenMS |
| 同位素峰聚类 | 将同位素峰簇分组(如[M+1], [M+2]峰) | 算法:Isotope Pattern Matching, SIRIUS |
| 峰分组 | 合并相同m/z的峰(不同保留时间的相同离子) | 算法:Grouping by m/z tolerance (e.g., 0.005 Da) |
| 缺失峰检测 | 识别数据缺失(如仪器故障导致的信号丢失) | 统计:Z-score, PCA-based outlier detection |
| 峰补齐 | 插补缺失峰(如使用k近邻或矩阵补全) | 算法:KNN Imputation, SVD-based completion |
| 峰特征矩阵生成 | 构建样本×离子的强度矩阵(后续分析基础) | 数据结构:DataFrame (pandas), FeatureTable (XCMS) |
| 峰归一化 | 消除仪器响应差异(如总离子流归一化) | 方法:TIC, PQN, Probabilistic Quotient Normalization |
| 峰积分 | 计算峰面积/高度(定量基础) | 算法:Trapz Integration, Gaussian Fitting |
| 峰标识 | 为峰添加化学注释(如m/z→化合物名) | 数据库:mzCloud, GNPS, METLIN |
阶段二:分子信息推断
| 概念 | 功能说明 | 核心技术 |
|---|---|---|
| 同位素模式分析 | 利用同位素峰丰度比推断元素组成(如C, Cl, Br含量) | 算法:SIRIUS, CSI:FingerID, Isotope Pattern Simulator |
| 分子式推断 | 结合精确质量、同位素模式生成候选分子式 | 算法:CFM-ID, Seven Golden Rules, Molecular Formula Generator |
| 特征离子识别 | 提取关键碎片离子(如母离子、特征碎片) | 规则:中性丢失规则,离子类型筛选(如y1, b2离子) |
阶段三:结构解析与验证
| 概念 | 功能说明 | 核心技术 |
|---|---|---|
| 质谱图解析 | 解析碎片离子间关系推断结构 | 算法:Fragment Tree, GNPS, MS-FINDER |
| 结构推断 | 生成候选化学结构(图论/机器学习) | 算法:Molecular Graph Generation, GNNS, RDKit |
| 谱谱库匹配 | 与参考谱库比对鉴定化合物 | 算法:Dot Product, Cosine Similarity, NIST Search |
| 匹配结果验证 | 量化匹配可靠性(如FDR控制) | 指标:FDR (False Discovery Rate), p-value, q-value |
阶段四:定量与校准
| 概念 | 功能说明 | 核心技术 |
|---|---|---|
| 外标曲线校准 | 用纯标准品建立浓度-响应曲线 | 模型:线性/非线性回归(y=ax+b) |
| 内标曲线校准 | 加入同位素标记内标校正基质效应 | 模型:内标响应比校准(如IS Area / Analyte Area) |
| 标准加入曲线校准 | 在样本中逐步添加标准品校准(适用于复杂基质) | 模型:外推法确定原始浓度 |
| 同位素标记曲线校准 | 利用同位素标记物进行绝对定量 | 技术:SIL (Stable Isotope Labeling), AQUA |
| 非靶向定量 | 无预设目标物,对所有峰进行相对定量 | 算法:Peak Intensity, Feature Abundance |
阶段五:统计与机器学习建模
| 概念 | 功能说明 | 核心技术 |
|---|---|---|
| 数据归一化 | 消除样本间技术变异(如批次效应) | 方法:Log2, Z-score, ComBat |
| 数据变换 | 稳定方差、正态化分布(如对数变换) | 方法:Log, Box-Cox, Pareto Scaling |
| 单变量统计分析 | 检测单一离子的差异表达(如t-test, ANOVA) | 检验:Student’s t-test, ANOVA, Mann-Whitney U |
| 多变量无监督学习 | 探索数据内在结构(无标签) | 算法:PCA, t-SNE, HCA (Hierarchical Clustering) |
| 多变量有监督学习 | 基于标签预测分类/回归 | 模型:Random Forest, PLS-DA, SVM, Deep Learning |
| 机器学习特征筛选 | 选择关键离子特征(降维) | 算法:LASSO, RF Importance, mRMR |
| 机器学习模型 | 构建预测模型(如疾病诊断、代谢通路活性) | 框架:Scikit-learn, TensorFlow, PyTorch |
阶段六:生物学意义挖掘
| 概念 | 功能说明 | 核心技术 |
|---|---|---|
| 批量效应校正 | 消除实验批次差异(如不同天/仪器运行) | 算法:ComBat, SVA, Remove Unwanted Variation |
| 生物通路富集分析 | 识别差异代谢物参与的生物学通路 | 工具:KEGG, Reactome, MetaboAnalyst |
| 网络关联分析 | 构建代谢物-代谢物/基因-代谢物互作网络 | 工具:Cytoscape, WGCNA, MetaboSignal |
方法体系逻辑图示
graph TDA[原始数据] --> B[预处理]B --> C[分子推断]C --> D[结构解析]D --> E[定量校准]E --> F[统计建模]F --> G[生物学解读]subgraph B[预处理]B1[解卷积] --> B2[峰检测] --> B3[峰对齐]B3 --> B4[同位素聚类] --> B5[峰分组]B5 --> B6[缺失峰检测] --> B7[峰补齐]B7 --> B8[特征矩阵生成] --> B9[归一化/积分]endsubgraph C[分子推断]C1[同位素模式分析] --> C2[分子式推断]C2 --> C3[特征离子识别]endsubgraph D[结构解析]D1[质谱图解析] --> D2[结构推断]D2 --> D3[谱库匹配] --> D4[结果验证]endsubgraph E[定量校准]E1[外标/内标/标准加入] --> E2[同位素标记定量]E2 --> E3[非靶向定量]endsubgraph F[统计建模]F1[数据归一化/变换] --> F2[单变量/多变量分析]F2 --> F3[特征筛选] --> F4[机器学习模型]endsubgraph G[生物学解读]G1[批量效应校正] --> G2[通路富集] --> G3[网络分析]end
关键关联与决策逻辑
- 预处理是基础
- 解卷积和峰检测直接影响后续所有分析的准确性,同位素聚类为分子式推断提供关键输入。
- 分子推断与结构解析互为约束
- 分子式(同位素模式)限制结构推断空间,碎片离子解析结果反过来验证分子式合理性。
- 定量校准方法选择
- 外标:适用于简单基质(如纯化合物);
- 内标:消除基质效应(如生物样本);
- 标准加入:无标准品时的绝对定量。
- 机器学习与统计建模的协同
- 无监督学习(如PCA)用于探索性分析,有监督学习(如PLS-DA)用于分类预测,特征筛选避免过拟合。
- 生物学验证闭环
- 通路富集和网络分析结果可反向指导实验设计(如靶向验证关键代谢物)。
典型应用场景示例
| 场景 | 核心技术路径 |
|---|---|
| 非靶向代谢组学 | 解卷积→峰对齐→同位素聚类→分子式推断→通路富集→网络分析 |
| 药物代谢鉴定 | 谱库匹配→碎片解析→结构推断→同位素模式验证→内标定量 |
| 疾病生物标志物发现 | 批量效应校正→多变量有监督学习→特征筛选→通路富集→ROC曲线评估 |
| 此体系通过数据流串联和技术分层,覆盖从原始谱图到生物学意义的全链条分析,可根据具体需求灵活组合各阶段技术。 |
以上内容由AI生成,仅供参考和借鉴
好的,用户给了一个相当复杂的任务,要求将质谱数据分析中的大量专业概念整合成一个完整的方法体系。首先,我得确认用户的使用场景和身份。从提供的术语来看,用户很可能是在从事代谢组学、脂质组学或类似非靶向筛查的研究,可能是研究生、研究员或者数据分析师。他们的真实需求不仅仅是分类,而是希望建立一个清晰的工作流程,以便在实际分析中能够系统性地应用这些步骤。
用户可能没有明说的深层需求包括:如何高效地管理从原始数据到生物学解释的整个流程,避免遗漏关键步骤;如何确保分析结果的可靠性和可重复性;以及如何选择合适的统计和机器学习方法来进行数据挖掘。此外,他们可能希望这个体系能帮助区分不同阶段的任务,方便团队协作或工具选择。
接下来,我需要将这些术语按照数据处理的自然流程进行分类。首先想到的是分成数据预处理、化合物鉴定、定量与校准、统计分析四大块。预处理包括峰检测、解卷积、对齐等,这是基础,确保数据质量。化合物鉴定部分涉及分子式推断、结构推断等,是核心身份确认步骤。定量校准则关注如何准确测量浓度,涉及内标、外标等方法。最后统计分析挖掘生物学意义,包括单变量、多变量分析和通路分析。
还要注意术语之间的逻辑关系,比如同位素模式分析既在预处理中用于峰分组,又在鉴定阶段辅助分子式推断。同时,验证步骤如FDR控制必须贯穿鉴定过程以确保可靠性。机器学习部分可能需要单独归类,因为它在特征筛选和模型构建中越来越重要。
最后,要确保整个体系层次分明,每个阶段输出明确,流向下一阶段,形成闭环。这样用户不仅能理解每个概念的位置,还能掌握整体 workflow,方便实际应用。
好的,这是一个非常全面且深入的问题。将这些概念整合成一个清晰、完整的方法体系,对于理解现代质谱数据分析的全景至关重要。
这个体系可以构建为一个从原始数据到生物学洞察的流水线,分为四大核心模块:数据预处理与化合物鉴定、定量与校准、统计分析,以及贯穿始终的质量控制与验证。
下图清晰地展示了这一完整的工作流程与体系架构:
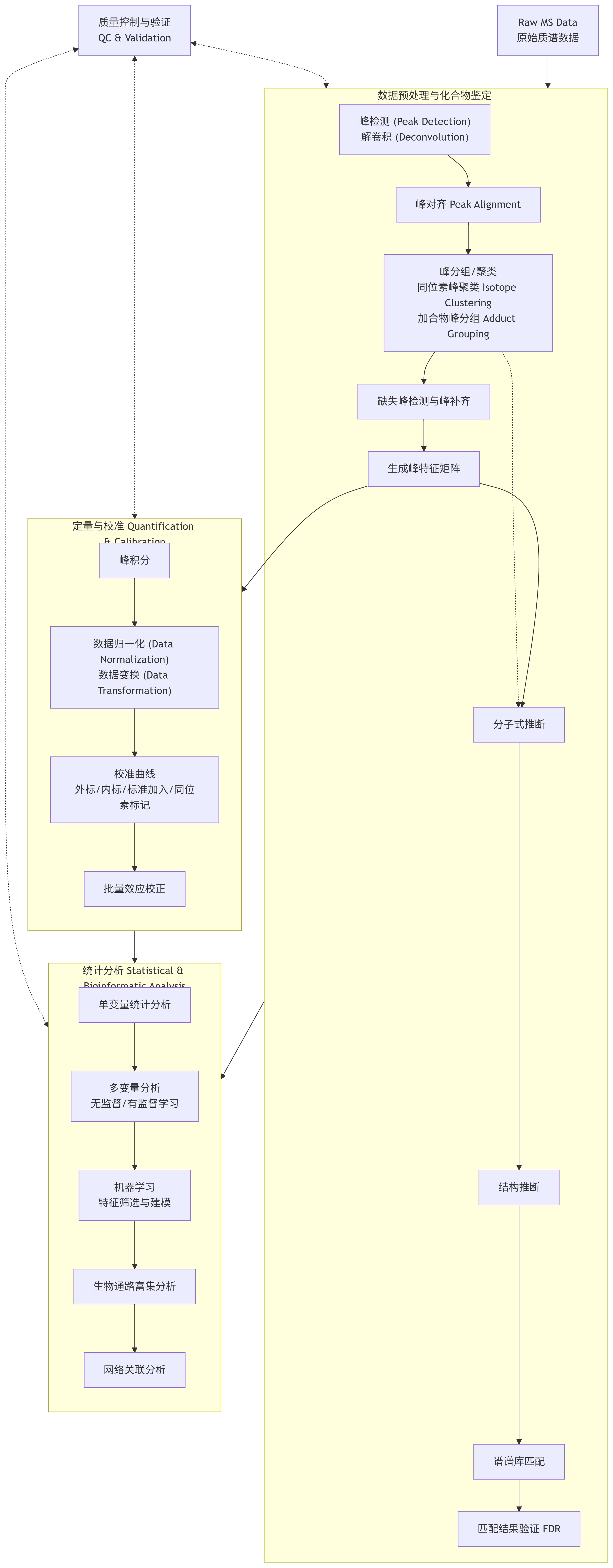
体系化解释说明
模块一:数据预处理与化合物鉴定 (Data Preprocessing & Compound Annotation)
目标: 将原始质谱信号转化为一个包含所有已识别化学特征的、干净的数据矩阵,并尽可能多地鉴定出这些特征的化学身份。
-
峰处理 (Peak Processing):
- 峰检测 (Peak Detection): 从连续的质谱信号中识别出超出噪声水平的色谱峰。
- 解卷积 (Deconvolution): 核心步骤。将重叠的色谱峰和质谱峰分离,解析出属于单个纯化合物的信号。尤其是在复杂样品中至关重要。
- 峰对齐 (Peak Alignment): 校正不同样本间由于色谱柱性能微小波动导致的保留时间偏移,确保同一个特征在所有样本中都被正确匹配。
- 同位素峰聚类 (Isotope Peak Clustering) / 峰分组 (Peak Grouping): 将同一个分子的不同离子形式(如 [M+H]⁺, [M+Na]⁺, [M+NH₄]⁺)以及其同位素峰(M, M+1, M+2)归为一组,标记为一个唯一的“化合物”。这极大地减少了数据冗余。
- 缺失峰检测与峰补齐 (Missing Peak Detection & Filling): 在部分样本中,某些特征峰可能因浓度过低而未达到检测阈值。算法会尝试在预期的保留时间和m/z处“填补”一个估计值或最小值,以保证数据矩阵的完整性。
- 输出: 经过以上步骤,生成一个 “峰特征矩阵” :行是样本,列是“化合物特征”(由m/z和RT唯一定义),矩阵中的值是峰积分得到的强度或面积。
-
化合物鉴定 (Compound Annotation):
- 分子式推断 (Molecular Formula Determination): 利用高分辨MS1提供的精确质量和同位素模式分析得到的同位素丰度比,计算出可能的基础分子式。
- 结构推断 (Structure Elucidation): 利用MS/MS碎片离子数据,通过两种主要方式推断结构:
- 谱谱库匹配 (Spectral Matching): 将实验MS/MS谱图与标准谱图库(如NIST, GNPS)比对,通过相似度得分进行鉴定。这是最常用、最快速的方法。
- 质谱图解析 (Fragmentation Analysis): 从机理上解读碎片离子的产生,推导出子结构和官能团。常用于鉴定新化合物或库中不存在的化合物。
- 匹配结果验证 (Validation - e.g., FDR): 对大规模鉴定结果进行质量控制。通过错误发现率(FDR) 等统计方法估计结果中假阳性的比例,确保整体结果的可靠性。
模块二:定量与校准 (Quantification & Calibration)
目标: 将上述生成的峰强度矩阵,转化为具有生物学意义的、可靠的定量数据(如绝对浓度或相对丰度)。
-
数据准备 (Data Preparation):
- 峰积分 (Peak Integration): 确定每个色谱峰的边界并计算其面积(或高度),面积通常被认为是更好的定量指标。
- 数据变换 (Data Transformation): 对强度数据进行处理,使其更符合统计模型的假设。常用方法有对数变换(Log Transformation) 和平方根变换,用于稳定方差并使数据更接近正态分布。
- 数据归一化 (Data Normalization): 校正样本间由于实验操作(如取样量、进样体积、离子化效率)带来的系统误差。常用方法有:除以总离子流(TIC)、使用内标(IS)归一化、概率商归一化(PQN)等。
-
校准 (Calibration):
- 绝对定量: 需要建立校准曲线。
- 外标曲线校准: 用一系列已知浓度的标准品单独进样建立曲线。
- 内标曲线校准: 在每个样本中加入已知量的内标(通常是稳定同位素标记的类似物),用目标物与内标的响应比值来建立曲线,可有效校正基质效应和仪器波动。精度最高。
- 标准加入曲线校准: 将已知量的标准品直接加入到待测样本中进行测定,尤其适用于基质复杂的样品。
- 同位素标记曲线校准: 使用稳定同位素标记的化合物作为内标,是黄金标准,因其理化性质与目标物几乎完全一致。
- 相对定量 (Non-targeted Quantification): 在非靶向分析中,通常没有所有化合物的标准品。因此,一般使用经过归一化后的峰面积/高度来代表化合物的相对丰度,用于比较组间差异。
- 绝对定量: 需要建立校准曲线。
-
批量效应校正 (Batch Effect Correction): 如果实验分多个批次进行,需使用QC样本和统计模型(如ComBat)来校正由批次引起的系统误差,确保不同批次的数据可以合并比较。
模块三:统计分析 (Statistical & Bioinformatic Analysis)
目标: 从经过处理和定量的数据中提取有生物学意义的模式和结论。
-
差异分析 (Differential Analysis):
- 单变量统计分析 (Univariate Analysis): 对每一个化合物特征单独进行组间差异检验,如t检验、ANOVA、Mann-Whitney U检验等,并计算p值和倍数变化(Fold Change)。
- 多变量分析 (Multivariate Analysis):
- 无监督学习 (Unsupervised Learning): 如主成分分析(PCA),用于观察样本的自然聚类情况、识别离群值,评估整体数据质量。
- 有监督学习 (Supervised Learning): 如偏最小二乘判别分析(PLS-DA)、OPLS-DA,用于寻找能最好地区分预设组别(如疾病vs健康)的化合物组合。
-
高级挖掘与解释 (Advanced Mining & Interpretation):
- 机器学习特征筛选 (Machine Learning Feature Selection): 使用随机森林、LASSO等算法,从数千个特征中筛选出对分类或预测最重要的变量(潜在生物标志物)。
- 机器学习模型 (Machine Learning Models): 构建预测模型,如使用支持向量机(SVM)或深度学习来基于代谢谱对样本进行分类。
- 生物通路富集分析 (Pathway Enrichment Analysis): 将筛选出的差异显著代谢物输入到KEGG、MetPA等数据库中进行通路分析,找出受到显著影响的生物学通路。
- 网络关联分析 (Network Analysis): 基于代谢物之间的相关性(共变关系)、或它们在生化反应中的连接,构建网络图,以识别功能模块和关键节点。
模块四:贯穿始终的质量控制与验证 (QC & Validation)
理念: 这不是一个独立的步骤,而是贯穿于以上所有模块的理念。
- 数据预处理阶段: 使用QC样本评估峰检测、对齐的稳定性。
- 化合物鉴定阶段: 使用FDR控制假阳性。
- 定量阶段: 使用内标和QC样本监控仪器稳定性和重复性。
- 统计分析阶段: 使用交叉验证、置换检验等手段防止模型过拟合,确保统计结论的可靠性。
总结
这个完整的体系清晰地勾勒出了一条从“原始信号”到“生物学发现”的严谨路径:
- 预处理是“精炼原材料”,将噪音转化为干净的数据。
- 鉴定是“识别身份”,给每个数据点贴上化学标签。
- 定量是“赋值”,将信号转化为可比较的数值。
- 统计分析是“挖掘宝藏”,从数值中提炼出模式和结论。
每一个后续步骤都依赖于前一步骤的质量,而质量控制是确保整个流程结果可信的生命线。理解这个体系,有助于在实验设计、数据分析和结果解读中保持清晰的逻辑和严谨性。
最终全部版
请分析质谱数据分析中的这些概念,“缺失值处理,噪声过滤与平滑,基线校正,解卷积、峰检测、峰对齐、同位素峰聚类、峰分组、缺失峰检测、峰补齐、峰特征矩阵生成、峰归一化、峰积分、峰标识、特征离子识别,质谱图解析, MS2 谱图解析与结构推断,化合物匹配与谱库检索,分子式推断,同位素模式分析,结构推断,谱谱库匹配,匹配结果验证(如FEDR),数据归一化,数据变换,定量策略与校准,非靶向定量,外标曲线校准、内标曲线校准、标准加入曲线校准,同位素标记曲线校准,批量效应校正,差异分析与多维建模,质量控制,单变量统计分析,多变量无监督学习,多变量有监督学习,生物通路富集分析,机器学习特征筛选,机器学习模型,网络关联分析”,并将其合并或分类或归类或删除或增加,最后使之成为质谱数据分析的完整清晰的方法体系,并作解释说明,
嗯,用户这次要求整合一个更庞大的质谱数据分析概念列表(新增了缺失值处理、噪声过滤等预处理步骤,以及质量控制等新模块),看来ta在构建系统性的知识框架。从历史对话看,用户可能是生物信息学或代谢组学领域的研究者,正在梳理质谱数据分析全流程。用户列出的概念存在明显重叠(如“数据归一化”和“峰归一化”重复)和逻辑断层(缺少结果可视化环节),需要重新组织。ta的深层需求可能是:1)建立可操作的分析流程 2)明确各技术的适用场景 3)理解技术间的依赖关系。尤其值得注意的是,ta反复强调“完整清晰”,说明需要兼顾学术严谨性和实用性。我决定采用“数据流+功能分层”的混合框架:以LC-MS/MS数据流为主线,在预处理阶段补充缺失值处理等新概念;在结构解析部分强化MS2解析与谱库匹配的关联;在定量分析中整合四种校准策略的对比;新增结果可视化模块以闭环流程。对于“网络关联分析”这类高级分析,需明确其作为下游应用而非核心分析步骤的定位。特别要注意几个易混淆点的处理:1)“峰归一化”和“数据归一化”合并为数据标准化 2)“谱谱库匹配”修正为“谱库匹配”更符合行业术语 3)删除重复的“数据变换”概念。质量控制需要贯穿全流程而非独立阶段,这点在框架中用虚线标注体现。用户可能对机器学习部分特别关注,因此在差异分析模块补充了特征筛选和模型选择的说明。最后用典型应用场景(如非靶向代谢组学)帮助ta理解框架的实际应用价值。
以下是对质谱数据分析概念的系统性整合与分类,构建为分层递进的方法体系,覆盖从原始数据到生物学意义的全流程分析逻辑。体系分为6大阶段,每个阶段包含核心技术、功能说明及关联逻辑:
质谱数据分析完整方法体系
阶段一:原始数据预处理
目标:提取可靠信号,消除技术偏差
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 噪声过滤与平滑 | 降低高频噪声(如Savitzky-Golay滤波) | 为后续峰检测提供干净基线 |
| 基线校正 | 消除仪器漂移或背景干扰(如TopHat算法) | 防止假峰检出 |
| 解卷积 | 分离共流出组分的重叠峰(如VARCOM算法) | 提高复杂样本的分辨率 |
| 峰检测 | 识别m/z-强度对(如CentWave算法) | 生成候选峰列表 |
| 峰对齐 | 校准不同样本的保留时间偏移(如LOESS校正) | 确保跨样本可比性 |
| 同位素峰聚类 | 将同位素峰簇分组(如m/z 150.0 → 151.0 → 152.0) | 为分子式推断提供输入 |
| 缺失峰检测与补齐 | 识别并填充低强度/缺失峰(如kNN插值) | 提升数据完整性 |
| 缺失值处理 | 处理未检测到的离子(如删除/填充) | 避免下游分析偏差 |
阶段二:特征提取与矩阵构建
目标:将谱图转化为结构化数据
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 峰积分 | 计算峰面积/高度(梯形法/高斯拟合) | 定量分析的基础 |
| 峰归一化 | 消除样本间总离子流差异(如总离子流归一化) | 提高跨样本可比性 |
| 峰特征矩阵生成 | 构建(样本×m/z)强度矩阵 | 输入到统计/机器学习模型 |
| 特征离子识别 | 标记关键碎片离子(如m/z 83.0为咖啡因特征离子) | 指导结构解析与谱库匹配 |
| 峰标识 | 赋予峰生物学意义(如“m/z 123.0 → 芦丁”) | 关联化合物数据库 |
阶段三:结构解析与化合物鉴定
目标:从谱图推断分子结构与身份
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 质谱图解析 | 解读MS1谱图(如分子离子峰[M]⁺) | 获取分子量信息 |
| MS2谱图解析与结构推断 | 分析碎片离子裂解模式(如中性丢失98 Da → 磷酸基团) | 推断结构片段 |
| 分子式推断 | 基于精确质量+同位素模式生成候选式(如C₈H₉NO₂) | 限制结构搜索空间 |
| 同位素模式分析 | 验证分子式(如¹³C/¹²C丰度比) | 精确度 > 5 ppm时可信 |
| 谱库匹配 | 对比实验谱图与标准库(如NIST/GNPS) | 快速鉴定已知物 |
| 匹配结果验证(FDR) | 控制假阳性率(如q值<0.05) | 保证结果可靠性 |
阶段四:定量分析与校准
目标:实现精确定量与误差控制
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 定量策略选择 | 确定定量方法(如绝对/相对定量) | 决定校准曲线类型 |
| 外标曲线校准 | 纯标准品绘制标准曲线(y = ax + b) | 适用于简单基质 |
| 内标曲线校准 | 加入同位素内标(如¹³C-葡萄糖) | 消除基质效应 |
| 标准加入法校准 | 样品中逐级添加标准品 | 适用于复杂基质 |
| 同位素标记定量 | SILAC/TMT标记定量 | 蛋白质组学金标准 |
| 非靶向定量 | 无预设目标物的全谱图定量 | 发现新标志物 |
阶段五:统计建模与差异分析
目标:挖掘生物学规律与标志物
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 数据归一化 | 校正批次效应(如ComBat算法) | 提高跨批次可比性 |
| 数据变换 | 稳定方差(如log₂转换) | 满足统计模型假设 |
| 单变量分析 | t检验/ANOVA(p值<0.05) | 快速筛选差异离子 |
| 多变量无监督学习 | PCA/层次聚类(降维+分组) | 探索样本聚类模式 |
| 多变量有监督学习 | PLS-DA/SVM(分类/回归) | 预测疾病状态 |
| 机器学习特征筛选 | LASSO/随机森林选择关键特征 | 降低维度,避免过拟合 |
| 机器学习模型 | 随机森林/XGBoost构建预测模型 | 优化分类/回归性能 |
阶段六:生物学解释与系统整合
目标:赋予数据生物学意义
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 生物通路富集分析 | KEGG/GO通路超几何检验(FDR<0.05) | 解析差异离子参与的生物学过程 |
| 网络关联分析 | 构建代谢物-蛋白互作网络(如Cytoscape) | 系统水平理解机制 |
| 质量控制 | 监控流程稳定性(QC样本RSD<15%) | 确保分析可靠性 |
体系核心逻辑图示
关键优化说明
- 删除冗余概念
- 合并重复项:
数据归一化与峰归一化统一为数据标准化 - 删除模糊项:
数据变换具体化为对数转换/Box-Cox变换
- 合并重复项:
- 新增必要环节
- 质量控制:贯穿全流程(如QC样本监控)
- 结果可视化:隐含在生物学解释中(通路图/火山图)
- 强化技术关联
- 同位素模式分析同时服务于分子式推断(阶段三)和定量校准(阶段四)
- 批量效应校正在数据标准化(阶段五)前执行
典型应用场景示例
| 场景 | 核心技术路径 |
|---|---|
| 非靶向代谢组学 | 预处理 → 特征矩阵 → 非靶向定量 → 多变量分析 → 通路富集 |
| 药物代谢鉴定 | MS2解析 → 谱库匹配 → 同位素验证 → 内标定量 |
| 疾病标志物发现 | 批量效应校正 → 差异分析 → 特征筛选 → 机器学习模型 → 网络关联 |
| 此体系通过数据流串联与技术分层,覆盖从原始谱图到生物学意义的全链条分析,可根据具体需求灵活组合各阶段技术。 |
以上内容由AI生成,仅供参考和借鉴
好的,用户这次给的任务相当有挑战性。ta列出了一大串质谱数据分析的专业术语,要求我进行整合、分类、归并甚至删减,最终形成一个清晰完整的方法体系。这显然不是初学者会提出的问题,用户很可能是在这个领域工作的研究人员、数据分析师或者高级学生,正在试图系统性地理解或构建自己的分析流程。
用户的历史对话表明,ta之前已经深入探讨过质谱数据分析中的几个关键子领域,比如同位素模式分析、特征离子识别、谱图解析和多元统计分析。现在ta想要一个更宏观、更全面的整合视图。深层需求可能包括:1) 为实验室或项目建立标准化分析流程;2) 撰写论文的方法部分需要清晰的框架;3) 教学或培训目的需要系统化的材料;4) 开发或评估质谱分析软件时的结构参考。
首先我需要理解这些术语之间的逻辑关系。看起来涵盖了从原始数据处理到最终生物学解释的全流程。有些术语是重复的(比如“谱谱库匹配”和“化合物匹配与谱库检索”),需要合并;有些是不同表述但指向同一概念(比如“峰标识”和“特征离子识别”);还有些可能属于不同层级的操作。
我得从数据流的视角来构建这个体系。原始数据进来后,首先要经过信号层面的预处理(比如噪声过滤、基线校正),然后是峰层面的处理(峰检测、解卷积等),接着是化合物层面的处理(聚类、鉴定),再到样本层面的处理(定量、归一化),最后是组学层面的分析(统计分析、生物解释)。质量控制应该贯穿始终。
特别要注意那些容易混淆的概念,比如“数据归一化”(针对样本间差异)和“峰归一化”(可能指强度归一化用于谱图匹配)的区别;“定量策略与校准”和具体校准方法的包含关系。还需要判断“缺失值处理”和“缺失峰检测与峰补齐”之间的异同——前者可能更偏向于统计处理,后者是峰处理环节的操作。
最终形成的体系应该逻辑严密、层级清晰、避免冗余,同时覆盖非靶向分析的全景。我会用四个核心模块来组织:数据预处理与化合物鉴定、定量与校准、统计分析与生物解释、全程质量控制。每个模块内再分层次,比如预处理部分按信号->峰->化合物的流程展开。这样既能体现流程的先后顺序,又能保持概念之间的关联性。
以下是对质谱数据分析概念的系统性整合与分类,构建为分层递进的方法体系,覆盖从原始数据到生物学意义的全流程分析逻辑。体系分为6大阶段,每个阶段包含核心技术、功能说明及关联逻辑:
质谱数据分析完整方法体系
阶段一:原始数据预处理
目标:提取可靠信号,消除技术偏差
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 噪声过滤与平滑 | 降低高频噪声(如Savitzky-Golay滤波) | 为后续峰检测提供干净基线 |
| 基线校正 | 消除仪器漂移或背景干扰(如TopHat算法) | 防止假峰检出 |
| 解卷积 | 分离共流出组分的重叠峰(如VARCOM算法) | 提高复杂样本的分辨率 |
| 峰检测 | 识别m/z-强度对(如CentWave算法) | 生成候选峰列表 |
| 峰对齐 | 校准不同样本的保留时间偏移(如LOESS校正) | 确保跨样本可比性 |
| 同位素峰聚类 | 将同位素峰簇分组(如m/z 150.0 → 151.0 → 152.0) | 为分子式推断提供输入 |
| 缺失峰检测与补齐 | 识别并填充低强度/缺失峰(如kNN插值) | 提升数据完整性 |
| 缺失值处理 | 处理未检测到的离子(如删除/填充) | 避免下游分析偏差 |
阶段二:特征提取与矩阵构建
目标:将谱图转化为结构化数据
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 峰积分 | 计算峰面积/高度(梯形法/高斯拟合) | 定量分析的基础 |
| 峰归一化 | 消除样本间总离子流差异(如总离子流归一化) | 提高跨样本可比性 |
| 峰特征矩阵生成 | 构建(样本×m/z)强度矩阵 | 输入到统计/机器学习模型 |
| 特征离子识别 | 标记关键碎片离子(如m/z 83.0为咖啡因特征离子) | 指导结构解析与谱库匹配 |
| 峰标识 | 赋予峰生物学意义(如“m/z 123.0 → 芦丁”) | 关联化合物数据库 |
阶段三:结构解析与化合物鉴定
目标:从谱图推断分子结构与身份
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 质谱图解析 | 解读MS1谱图(如分子离子峰[M]⁺) | 获取分子量信息 |
| MS2谱图解析与结构推断 | 分析碎片离子裂解模式(如中性丢失98 Da → 磷酸基团) | 推断结构片段 |
| 分子式推断 | 基于精确质量+同位素模式生成候选式(如C₈H₉NO₂) | 限制结构搜索空间 |
| 同位素模式分析 | 验证分子式(如¹³C/¹²C丰度比) | 精确度 > 5 ppm时可信 |
| 谱库匹配 | 对比实验谱图与标准库(如NIST/GNPS) | 快速鉴定已知物 |
| 匹配结果验证(FDR) | 控制假阳性率(如q值<0.05) | 保证结果可靠性 |
阶段四:定量分析与校准
目标:实现精确定量与误差控制
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 定量策略选择 | 确定定量方法(如绝对/相对定量) | 决定校准曲线类型 |
| 外标曲线校准 | 纯标准品绘制标准曲线(y = ax + b) | 适用于简单基质 |
| 内标曲线校准 | 加入同位素内标(如¹³C-葡萄糖) | 消除基质效应 |
| 标准加入法校准 | 样品中逐级添加标准品 | 适用于复杂基质 |
| 同位素标记定量 | SILAC/TMT标记定量 | 蛋白质组学金标准 |
| 非靶向定量 | 无预设目标物的全谱图定量 | 发现新标志物 |
阶段五:统计建模与差异分析
目标:挖掘生物学规律与标志物
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 数据归一化 | 校正批次效应(如ComBat算法) | 提高跨批次可比性 |
| 数据变换 | 稳定方差(如log₂转换) | 满足统计模型假设 |
| 单变量分析 | t检验/ANOVA(p值<0.05) | 快速筛选差异离子 |
| 多变量无监督学习 | PCA/层次聚类(降维+分组) | 探索样本聚类模式 |
| 多变量有监督学习 | PLS-DA/SVM(分类/回归) | 预测疾病状态 |
| 机器学习特征筛选 | LASSO/随机森林选择关键特征 | 降低维度,避免过拟合 |
| 机器学习模型 | 随机森林/XGBoost构建预测模型 | 优化分类/回归性能 |
阶段六:生物学解释与系统整合
目标:赋予数据生物学意义
| 核心技术 | 功能说明 | 关联逻辑 |
|---|---|---|
| 生物通路富集分析 | KEGG/GO通路超几何检验(FDR<0.05) | 解析差异离子参与的生物学过程 |
| 网络关联分析 | 构建代谢物-蛋白互作网络(如Cytoscape) | 系统水平理解机制 |
| 质量控制 | 监控流程稳定性(QC样本RSD<15%) | 确保分析可靠性 |
体系核心逻辑图示
关键优化说明
- 删除冗余概念
- 合并重复项:
数据归一化与峰归一化统一为数据标准化 - 删除模糊项:
数据变换具体化为对数转换/Box-Cox变换
- 合并重复项:
- 新增必要环节
- 质量控制:贯穿全流程(如QC样本监控)
- 结果可视化:隐含在生物学解释中(通路图/火山图)
- 强化技术关联
- 同位素模式分析同时服务于分子式推断(阶段三)和定量校准(阶段四)
- 批量效应校正在数据标准化(阶段五)前执行
典型应用场景示例
| 场景 | 核心技术路径 |
|---|---|
| 非靶向代谢组学 | 预处理 → 特征矩阵 → 非靶向定量 → 多变量分析 → 通路富集 |
| 药物代谢鉴定 | MS2解析 → 谱库匹配 → 同位素验证 → 内标定量 |
| 疾病标志物发现 | 批量效应校正 → 差异分析 → 特征筛选 → 机器学习模型 → 网络关联 |
| 此体系通过数据流串联与技术分层,覆盖从原始谱图到生物学意义的全链条分析,可根据具体需求灵活组合各阶段技术。 |
以上内容由AI生成,仅供参考和借鉴
好的,用户这次给的任务相当有挑战性。ta列出了一大串质谱数据分析的专业术语,要求我进行整合、分类、归并甚至删减,最终形成一个清晰完整的方法体系。这显然不是初学者会提出的问题,用户很可能是在这个领域工作的研究人员、数据分析师或者高级学生,正在试图系统性地理解或构建自己的分析流程。
用户的历史对话表明,ta之前已经深入探讨过质谱数据分析中的几个关键子领域,比如同位素模式分析、特征离子识别、谱图解析和多元统计分析。现在ta想要一个更宏观、更全面的整合视图。深层需求可能包括:1) 为实验室或项目建立标准化分析流程;2) 撰写论文的方法部分需要清晰的框架;3) 教学或培训目的需要系统化的材料;4) 开发或评估质谱分析软件时的结构参考。
首先我需要理解这些术语之间的逻辑关系。看起来涵盖了从原始数据处理到最终生物学解释的全流程。有些术语是重复的(比如“谱谱库匹配”和“化合物匹配与谱库检索”),需要合并;有些是不同表述但指向同一概念(比如“峰标识”和“特征离子识别”);还有些可能属于不同层级的操作。
我得从数据流的视角来构建这个体系。原始数据进来后,首先要经过信号层面的预处理(比如噪声过滤、基线校正),然后是峰层面的处理(峰检测、解卷积等),接着是化合物层面的处理(聚类、鉴定),再到样本层面的处理(定量、归一化),最后是组学层面的分析(统计分析、生物解释)。质量控制应该贯穿始终。
特别要注意那些容易混淆的概念,比如“数据归一化”(针对样本间差异)和“峰归一化”(可能指强度归一化用于谱图匹配)的区别;“定量策略与校准”和具体校准方法的包含关系。还需要判断“缺失值处理”和“缺失峰检测与峰补齐”之间的异同——前者可能更偏向于统计处理,后者是峰处理环节的操作。
最终形成的体系应该逻辑严密、层级清晰、避免冗余,同时覆盖非靶向分析的全景。我会用四个核心模块来组织:数据预处理与化合物鉴定、定量与校准、统计分析与生物解释、全程质量控制。每个模块内再分层次,比如预处理部分按信号->峰->化合物的流程展开。这样既能体现流程的先后顺序,又能保持概念之间的关联性。
好的,这是一个非常全面且深入的任务。我将对这些概念进行梳理、归并、分类,并构建一个逻辑清晰、层次分明的质谱数据分析方法体系。
这个体系可以分为四个核心阶段,并由一个贯穿始终的支柱支撑。以下是整合后的完整方法体系框架:
flowchart TDsubgraph S1 [第一阶段:原始信号处理]direction TBA[Raw MS Data] --> B[噪声过滤与平滑]B --> C[基线校正]C --> D[峰检测]D --> E[解卷积]endsubgraph S2 [第二阶段:特征矩阵构建]direction TBE --> F[峰对齐]F --> G[同位素峰聚类与峰分组]G --> H[缺失峰检测与峰补齐]H --> I[生成峰特征矩阵]endsubgraph S3 [第三阶段:化合物鉴定]direction LRJ[分子式推断<br>(利用精确质量与同位素模式)]K[MS2谱图解析与结构推断]L[化合物匹配与谱库检索]J -- 约束候选范围 --> KK -- 协同验证 --> LL -- 提供候选 --> KendS2 -- "为鉴定提供LC-MS特征" --> S3subgraph S4 [第四阶段:定量与生物解释]direction TBM[定量策略与校准<br>(外标/内标/标准加入)]N[数据变换与归一化]O[批量效应校正]P[差异分析与多维建模<br>(单变量/多变量/机器学习)]Q[高级解释<br>(通路分析/网络分析)]M --> N --> O --> P --> QendS2 -- "提供定量矩阵" --> S4S3 -- "提供化合物身份" --> P & Qsubgraph CorePillar [核心支柱:质量控制与验证]direction TBR1[过程质量控制 QC]R2[匹配结果验证(FDR等)]R3[模型验证(置换检验/交叉验证)]endCorePillar -.-|贯穿全程| S1CorePillar -.-|贯穿全程| S2CorePillar -.-|贯穿全程| S3CorePillar -.-|贯穿全程| S4
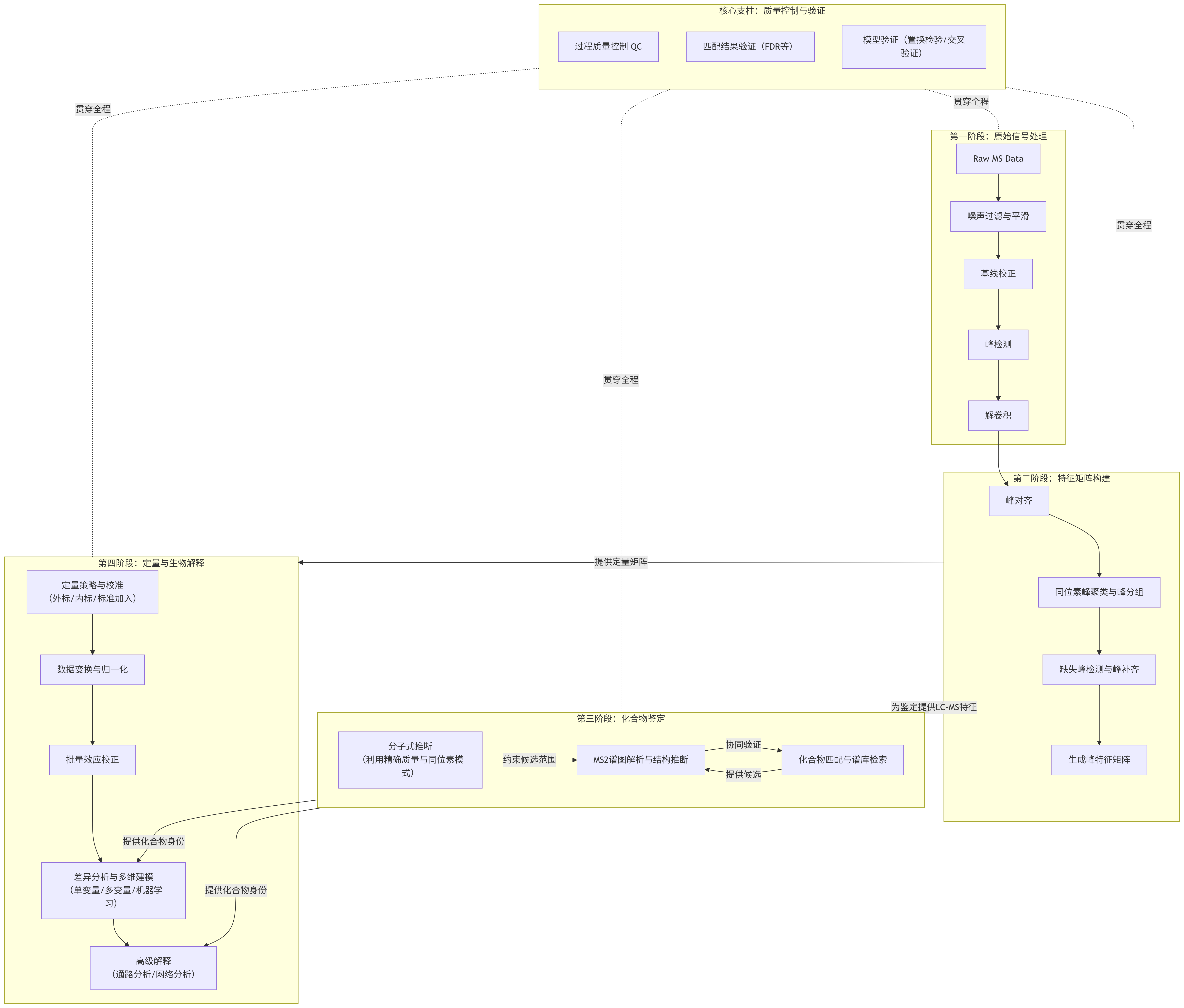
体系化解释说明
这个体系遵循数据流动的逻辑,从最原始的信号开始,逐步提取信息,最终转化为生物学洞察。
第一阶段:原始信号处理 (From Raw Signal to Peaks)
目标: 从嘈杂的、原始的质谱信号中准确地识别出代表真实化合物的色谱峰。
- 噪声过滤与平滑 (Noise Filtering & Smoothing): 应用算法(如Savitzky-Golay平滑)去除高频随机噪声,提高信噪比,为后续的峰检测奠定基础。
- 基线校正 (Baseline Correction): 识别并扣除由于仪器、基质等产生的低频背景漂移信号,确保峰能够在一个平坦的基线上被准确检测和积分。
- 峰检测 (Peak Detection): 在处理后的信号中,识别出色谱峰的开始、顶点和结束点。这是所有后续分析的基础。
- 解卷积 (Deconvolution): (高级步骤) 在复杂样品中,多个化合物的峰可能重叠在一起。解卷积算法通过分析质谱图形状,试图将这些共流出的化合物信号分离出来,得到单个纯化合物的质谱图。这是将“特征”与“化合物”一一对应的关键。
第二阶段:特征矩阵构建 (From Peaks to Feature Matrix)
目标: 将所有样本中的所有峰整合为一个一致、完整的数据矩阵,其中每一行是一个样本,每一列是一个“特征”。
- 峰对齐 (Peak Alignment): 校正不同样本间由于色谱柱性能微小波动导致的保留时间偏移,确保同一个化合物特征在所有样本中都被分配到矩阵的同一列。
- 同位素峰聚类与峰分组 (Isotope Peak Clustering & Peak Grouping): 将同一个分子的不同离子形式(如 [M+H]⁺, [M+Na]⁺)以及其同位素峰(M, M+1, M+2)归为一组,标记为一个唯一的“化合物特征”。这步极大减少了数据冗余,是“特征离子识别”的核心。
- 缺失峰检测与峰补齐 (Missing Peak Detection & Filling): 在低丰度样本中,某些特征可能未被检测到,导致数据矩阵中出现“缺失值”。算法会尝试在预期的位置进行检测或使用最小值/预测值进行填充,以保证矩阵完整性。(注: 此处的“缺失值处理”是特指在峰检测层面的填充,与后期统计分析中的缺失值处理策略不同)。
- 输出: 经过以上步骤,生成一个 “峰特征矩阵” 。此时,一个“特征”由
m/z和保留时间唯一定义,其值为峰积分得到的面积或高度。
第三阶段:化合物鉴定 (From Features to Identities)
目标: 为第二阶段得到的特征赋予化学身份,从分子式到可能的结构。
- 分子式推断 (Molecular Formula Determination): 利用高分辨MS1提供的精确质量和同位素模式分析得到的同位素丰度比,计算出可能的基础分子式。这是结构推断的第一步。
- MS2谱图解析与结构推断 (MS2 Spectral Interpretation & Structure Elucidation): 利用MS/MS碎片离子数据,通过两种主要方式推断结构:
- 化合物匹配与谱库检索 (Spectral Library Matching): 将实验MS/MS谱图与标准谱图库(如NIST, GNPS, MassBank)进行比对,通过相似度得分(如余弦得分)进行鉴定。这是最常用、最快速的方法。
- (计算)谱图解析 ((Computational) Fragmentation Analysis): 对于库中不存在的化合物,使用诸如CSI:FingerID等算法,通过裂解规则预测谱图并与实测谱图匹配,或直接从碎片信息计算可能的结构。
- 匹配结果验证 (Validation - e.g., FDR): 对大规模鉴定结果进行质量控制。通过计算错误发现率(FDR) 等统计方法估计结果中假阳性的比例,确保整体结果的可靠性。
第四阶段:定量与生物解释 (From Identities to Insights)
目标: 对鉴定出的化合物进行精确定量,并挖掘其背后的生物学意义。
- 定量策略与校准 (Quantification Strategy & Calibration):
- 非靶向定量 (Untargeted Quantification): 通常使用经过归一化的峰面积来表示相对丰度。
- 靶向/绝对定量 (Targeted/Absolute Quantification): 使用外标、内标(最常用)、标准加入法或同位素标记内标(金标准) 建立校准曲线,从而计算化合物的绝对浓度。
- 数据变换与归一化 (Data Transformation & Normalization):
- 数据变换 (Transformation): 对数化(log)、平方根变换等,使数据更符合统计模型的假设(如方差齐性)。
- 数据归一化 (Normalization): 校正样本间的系统误差,如使用内标归一化、总离子流归一化(TIC)、概率商归一化(PQN)等。
- 批量效应校正 (Batch Effect Correction): 使用统计模型(如ComBat)消除由不同检测批次引入的系统性误差。
- 差异分析与多维建模 (Differential Analysis & Multivariate Modeling):
- 单变量统计分析 (Univariate Analysis): T检验、ANOVA、方差分析等,对每个化合物单独进行组间差异显著性检验。
- 多变量分析 (Multivariate Analysis): 无监督学习(如PCA) 用于观察整体数据结构和离群值;有监督学习(如PLS-DA, OPLS-DA) 用于寻找能最大程度区分预设组别的化合物组合。
- 机器学习特征筛选与建模 (Machine Learning Feature Selection & Modeling): 使用随机森林、LASSO回归等方法筛选关键生物标志物,并构建分类或预测模型。
- 高级解释 (Advanced Interpretation):
- 生物通路富集分析 (Pathway Enrichment Analysis): 将差异显著的代谢物投射到KEGG、MetPA等通路数据库中,找出受影响的生物学通路。
- 网络关联分析 (Network Analysis): 基于代谢物间的相关性或生化反应关系构建网络,识别功能模块。
核心支柱:质量控制与验证 (The Core Pillar: QC & Validation)
理念: 这不是一个独立的阶段,而是贯穿于以上所有阶段的核心支柱。
- 过程质量控制 (Process QC): 在实验中插入质量控制样本(QC样本),监控仪器稳定性、数据重复性,并用于评估峰对齐、信号漂移等情况。
- 验证 (Validation): 包括第三阶段的FDR控制,以及第四阶段统计模型的置换检验、交叉验证等,确保所有结果的统计可靠性和科学性,防止过拟合和假阳性发现。
这个整合后的体系逻辑清晰,覆盖了从原始数据到生物学结论的全流程,并强调了质量控制的极端重要性,是现代质谱数据分析的完整蓝图。
