差异基因散点图绘制教程
差异基因散点图绘制教程
本期教程
小杜的生信笔记,自2021年11月开始做的知识分享,主要内容是R语言绘图教程、转录组上游分析、转录组下游分析等内容。凡事在社群同学,可免费获得自2021年11月份至今全部教程,教程配备事例数据和相关代码,我们会持续更新中。
往期教程部分内容





绘图教程
导入所需R包
if (!require('ggplot2')) install.packages('ggplot2'); library('ggplot2')
if (!require('DESeq2')) install.packages('DESeq2'); library('DESeq2')
if (!require('biomaRt')) install.packages('biomaRt'); library('biomaRt')
if (!require('GEOquery')) install.packages('GEOquery'); library('GEOquery')
if (!require('vsn')) install.packages('vsn'); library('vsn')
if (!require('pheatmap')) install.packages('pheatmap'); library('pheatmap')
导入数据
cts <- read.csv("Input.data.csv",header = T, row.names = 1)
差异分析
##'@分组
sample_type = gsub(".*P7ma","Quiescent",gsub(".*P6ma","Cycling",colnames(cts)));
coldata = data.frame(row.names=colnames(cts), phenotype = sample_type ); #'@DESeq2差异分析
ddsMF <- DESeqDataSetFromMatrix(countData = cts, colData = coldata, design = ~ phenotype)
ddsMF <- DESeq(ddsMF)
resultsNames(ddsMF) #'get normalized counts'@标准化数据
nds <- DESeqTransform( SummarizedExperiment(log2(counts(ddsMF, normalized=TRUE) + 1), colData=colData(ddsMF)) ) # this gives log2(norm_cts + 1)
vsd <- vst(ddsMF, blind=FALSE) # variance stabilization
创建data.frame数据矩阵
quiescent = rownames(coldata)[coldata$phenotype=="Quiescent"]
cycling = rownames(coldata)[coldata$phenotype=="Cycling"]
data_cts_mets_avg = data.frame("quiescent"=rowMeans(assay(vsd)[, quiescent],na.rm=T),"cycling"=rowMeans(assay(vsd)[, cycling],na.rm=T))
绘图
##'@显著性差异基因
upCQ = resCQ[!is.na(resCQ$padj) & resCQ$padj < 0.1 & resCQ$log2FoldChange > 1,]
dwCQ = resCQ[!is.na(resCQ$padj) & resCQ$padj < 0.1 & resCQ$log2FoldChange < -1,]
dim(upCQ)
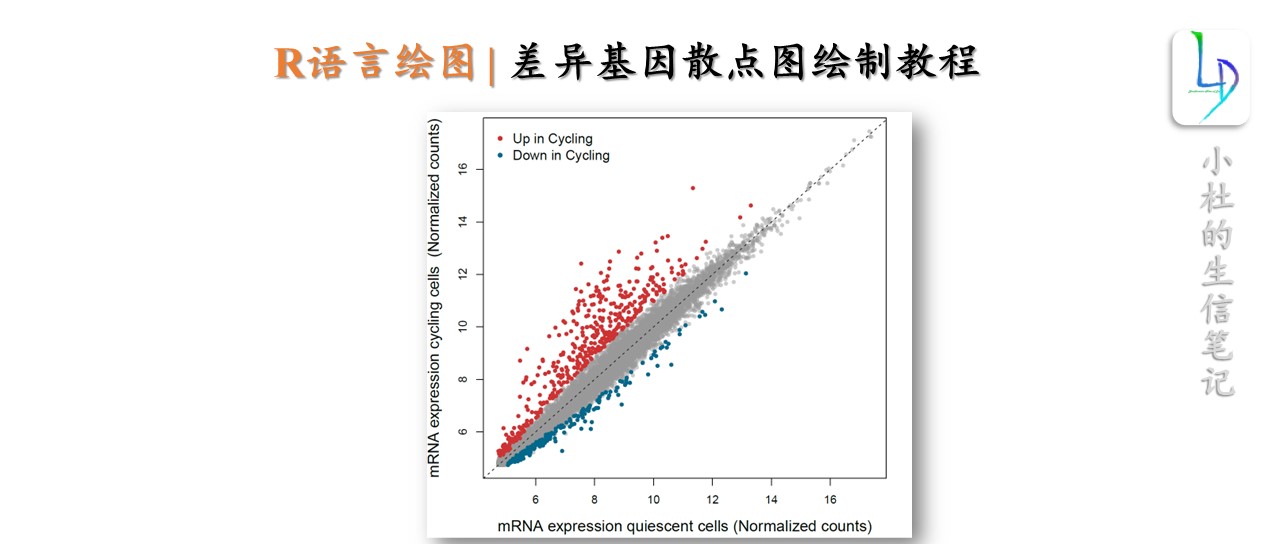
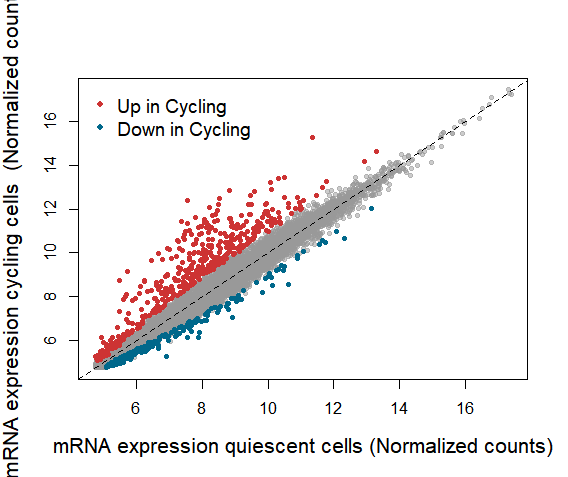
dim(dwCQ)plot(data_cts_mets_avg$quiescent, data_cts_mets_avg$cycling, pch=20, xlab="mRNA expression quiescent cells (Normalized counts)", ylab="mRNA expression cycling cells (Normalized counts)",cex.lab=1.3,col=rgb(0.6,0.6,0.6,0.5));
abline(0,1,lty=2,col="black");
points(data_cts_mets_avg[rownames(upCQ),"quiescent"], data_cts_mets_avg[rownames(upCQ),"cycling"],pch=20,col=colors()[c(35)])
points(data_cts_mets_avg[rownames(dwCQ),"quiescent"], data_cts_mets_avg[rownames(dwCQ),"cycling"],pch=20,col=colors()[c(125)])
legend("topleft",legend=c("Up in Cycling","Down in Cycling"),col=colors()[c(35,125)],pch=20,inset=0.01,bty="n",cex=1.2)
若我们的教程对你有所帮助,请
点赞+收藏+转发,这是对我们最大的支持。
差异基因散点图绘制教程
往期部分文章
1. 最全WGCNA教程(替换数据即可出全部结果与图形)
-
WGCNA分析 | 全流程分析代码 | 代码一
-
WGCNA分析 | 全流程分析代码 | 代码二
-
WGCNA分析 | 全流程代码分享 | 代码三
-
WGCNA分析 | 全流程分析代码 | 代码四
-
WGCNA分析 | 全流程分析代码 | 代码五(最新版本)
2. 精美图形绘制教程
- 精美图形绘制教程
3. 转录组分析教程
-
转录组上游分析教程[零基础]
-
一个转录组上游分析流程 | Hisat2-Stringtie
4. 转录组下游分析
-
批量做差异分析及图形绘制 | 基于DESeq2差异分析
-
GO和KEGG富集分析
-
单基因GSEA富集分析
-
全基因集GSEA富集分析
小杜的生信筆記 ,主要发表或收录生物信息学教程,以及基于R分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!
