TCGA数据下载推荐:R语言easyTCGA包
#使用easyTCGA获取数据
#清空
rm(list=ls())
gc()
# 安装bioconductor上面的R包
options(BioC_mirror="https://mirrors.tuna.tsinghua.edu.cn/bioconductor")
if(!require("BiocManager")) install.packages("BiocManager")
if(!require("TCGAbiolinks")) BiocManager::install("TCGAbiolinks")
if(!require("SummarizedExperiment")) BiocManager::install("SummarizedExperiment")
if(!require("DESeq2")) BiocManager::install("DESeq2")
if(!require("edgeR")) BiocManager::install("edgeR")
if(!require("limma")) BiocManager::install("limma")
# 安装cran上面的R包
if(!require("survival")) install.packages("survival")
if(!require("broom")) install.packages("broom")
if(!require("devtools")) install.packages("devtools")
if(!require("cli")) install.packages("cli")
#devtools::install_github("ayueme/easyTCGA")
library(easyTCGA)
help(package="easyTCGA")
setwd("F:\\TCGA\\TCGA-COAD")
#下载mRNA、lncRNA和临床信息
COAD<-getmrnaexpr("TCGA-COAD")#原始下载的count, TPM, FPKM 均没有经过log2转化
#下载miRNA
COAD_miRNA<-getmirnaexpr("TCGA-COAD")
#下载copy number variation data
COAD_cnv<-getcnv("TCGA-COAD")
#下载masked somatic mutation 体细胞突变
COAD_snv<-getsnvmaf("TCGA-COAD")
#下载DNA methylation beta value 甲基化数据
getmethybeta("TCGA-COAD")
#从下载目录中打开数据
#差异分析
diff<-diff_analysis(exprset=mrna_expr_counts,#没有经过log2转化project="TCGA-COAD",save=F)#批量生存分析
surv<-batch_survival(exprset=mrna_expr_counts,clin=clin_info,is_count = T,optimal_cut = TRUE,project="TCGA-COAD",save_data = FALSE,min_sample_size = 5,print_index = TRUE
)
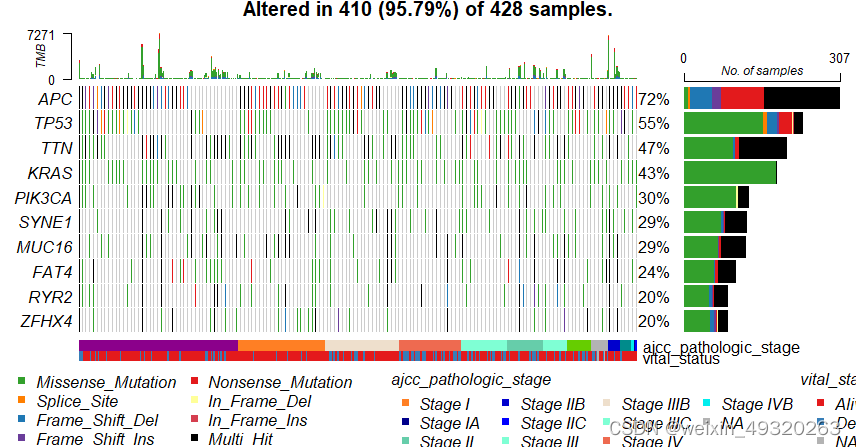
#突变分析:瀑布图
#BiocManager::install("maftools")
library(maftools)
maf<-read.maf(snv,clinicalData=clin_snv)
plotmafSummary(maf)
colnames(clin_snv)
oncoplot(maf=maf,clinicalFeatures=c("ajcc_pathologic_stage","vital_status"),top=10,sortByAnnotation=T
)

#绘制KM曲线
dim(mrna_expr_counts)
set.seed(123)
colnames(clin_info)
clin<-data.frame(time=clin_info$days_to_last_follow_up,event=clin_info$vital_status)
clin$event<-ifelse(clin$event=="Alive",0,1)
plot_KM(exprset=mrna_expr_counts, marker="CHPF", #基因clin=clin, optimal_cut = TRUE, return_data = TRUE)
#正常和癌症组织基因表达对比箱线图
rownames(mrna_expr_counts)
plot_gene_paired(exprset=mrna_expr_counts, marker="CHPF", #基因return_data = TRUE)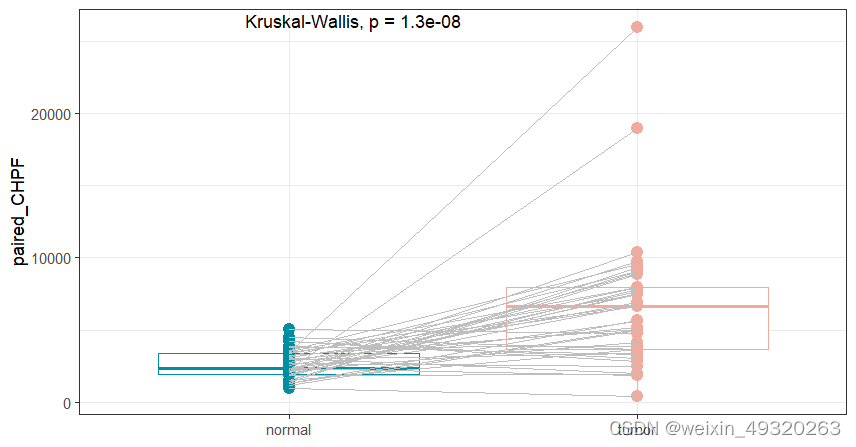
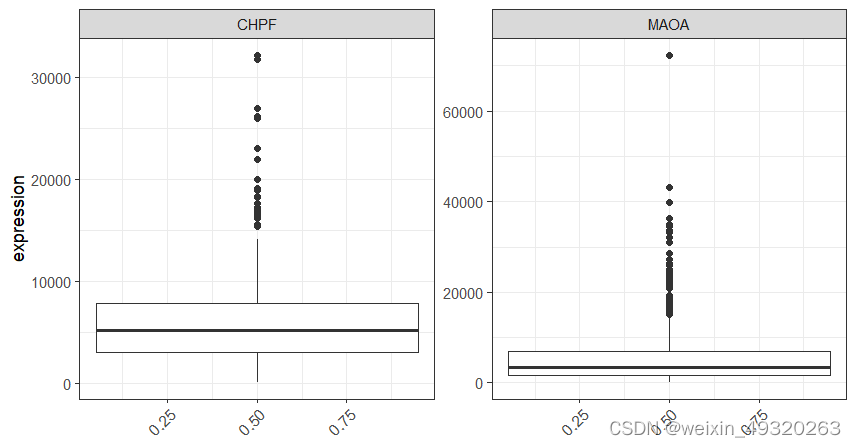
#比较组间基因表达差异
set.seed(123)
group=sample(c(0,1),524,replace = T)
plot_gene(exprset=mrna_expr_counts, marker=c("CHPF","MAOA"), group=group, return_data = TRUE)